
ПРОИСХОЖДЕНИЕ СЛАВЯН,

Предваряя главу таким названием, автор вовсе не имеет в виду, что все работы в области популяционной генетики ошибочные и совершенно бездарные. Нет, если они выполнены правильно, и вопросы поставлены правильные, и методология правильная, и альтернативные варианты ответов рассмотрены и последовательно и обосновано отведены, то с ними, этими работами, и их авторами все в порядке.
Беда в том, что такие работы мне лично пока не попадались, если они относились к рассмотрению древних миграций человека, истории популяций, датировок древних (или не очень древних) популяций, и даже корректных характеристик популяций, хотя уж последнее-то популяционные генетики должны уметь делать.
Видимо, либо статьи с корректным анализом популяционных генетиков мне как-то не попадались, хотя я их читаю десятками, либо у меня другие представления о понятии «корректный анализ». И дело не во мне лично, поскольку мои коллеги с подобным естественнонаучным образованием, как и у меня, пришли к тому же мнению относительно современного круга популяционных генетиков. Что-то системное с ними, современными попгенетиками, с уровнем их мышления, их школой, их ценностями в науке.
На днях коллега по международному форуму RootsWeb в полушутливой (но и в полусерьезной) форме предложил мне не нарушать традиций популяционной генетики (в том, что они называют «генетической генеалогией») и не требовать от попгенетиков доказательств выдвигаемых ими положений (в том случае это касалось Чингисхана, которому совершенно бездоказательно приписывают гаплотип, который якобы в больших количествах распространен по Евразии). Он сформулировал это в следующем виде: «The tradition requires to accept claims without evidence. Without this tradition the field of Genetic Genealogy couldn't exist. It's when you have evidence, then you have a problem. Evidence makes it is virtually impossible for a proposal to even get considered. So Genghis Khan, it is. Period. «Everyone knows that». There is no evidence around to complicate things».
В переводе это звучит примерно так: «Традиция (популяционной генетики) требует принимать положения без каких-либо доказательств. Без этой традиции область генетической генеалогии не может существовать. Напротив, когда у вас есть доказательства, вы создаете себе проблемы, Доказательства делают ваши положения практически невозможными для рассмотрения (попгенетиками). Так что (это гаплотип) Чингисхана, Точка, «Любой это знает». То, что к этому нет доказательств, делает ситуацию значительно проще и понятней».
Шутки шутками, но устойчивая неспособность (или нежелание, или отсутствие добротной академической школы) к рассмотрению альтернативных вариантов гипотез является яркой отличительной особенностью современной популяционной генетики (точнее, современных популяционных генетиков) как в России, так и в других странах. Поражает примитивность «академических» статей попгенетиков, выхватывание одного какого-то фактора из совокупности многих, подгонка выводов под устоявшиеся положения, страх к свежим выводам и новым концепциям. Гаплотипы, при всем богатстве информации, в них заключенной, даже не рассматриваются. Поразительно, но во всей объемной книге Балановских «Русский генофонд на Русской равнине» (2007, 416 с.) не рассмотрено ни одного (!) гаплотипа. Это — современная популяционная генетика. И это при том, что к году выхода книги гаплотипы уже вовсю изучались и публиковались по меньшей мере десяток лет. Иначе говоря, авторы шли на публикацию заведомо устаревшей книги.
Устойчивая настороженность к подходам и выводам современной популяционной генетики у автора настоящей статьи появилась с самого начала возникновения их «теорий». Оказалось, что их «теории» и «выводы» очень часто основаны практически ни на чем. На чем, например, был основан вывод, за который попгенетики держались много лет — что носители гаплогруппы R1b якобы жили в Европе 30 тыс. лет назад? Я скажу, на чем — на том, что люди в Европе действительно жили в те времена. И они были просто «с потолка» отнесены к гаплогруппе R1b. Почему, на каком основании? Да на том, что они СЕЙЧАС там живут. И потому что в Европе 30 тыс. лет назад жили кроманьонцы. А почему тогда именно гаплогруппа R1b? А потому. Потому что и сейчас там живут, наряду, правда, с десятком других гаплогрупп. Но те какие-то несимпатичные.
А поскольку попгенетики обожают «теорию палеолитической непрерывности» без ее критического осмысления, то из нее следует вывод, характерный для уровня мышления попгенетиков — раз живут сейчас, значит, жили раньше, и 30 тыс. лет назад. Непрерывность же, не так ли?
Кроме этого примитивного положения, НИ ЕДИНОГО основания к этим пресловутым 30 тыс. лет для гаплогруппы R1b в Европе выдвинуто не было — в том числе «отцами-основателями» — Хаммером, Семино (мать-основатель), Уэллсом и другими. Вот, например, цитата из книги Уэллса (Spencer Wells, «Deep Ancestry», 2006): «Around 30,000 years ago, a descendant of the clan making its way into Europe gave rise to marker M343, the defining marker of haplogroup R1b. These travelers are direct descendants of the people who dominated the human expansion into Europe, the Cro-Magnon». To есть, что 30 тыс. лет назад образовалась гаплогруппа R1b, прямой потомок европейских кроманьонов. На основании чего получена датировка 30 тыс. лет назад для гаплогруппы R1b в Европе, и что якобы она была потомком кроманьонского человека? Анализировались гаплотипы носителей гаплогруппы R1b в Европе? Считались и рассчитывались мутации в гаплотипах? Проводились соответствующие экстраполяции? Нашли ископаемые гаплотипы R1b в Европе? Ничего этого и близко не было. Это — беспардонная ложь, если говорить напрямую. Цифра просто была придумана, и вошла в справочники. Придумано и про потомков кроманьонцев. Это — популяционная генетика, ее методология. На самом деле расчеты по гаплотипам гаплогруппы R1b в Европе показывают, что гаплогруппа появилась в Европе в разных местах в интервале от 4800 до 4500 лет назад, видимо, прибыв по нескольким направлениям, все исходно из Азии. Никаких R1b среди кроманьонцев отродясь не было.
Откуда у попгенетиков выводы, повторяемые много раз, что гаплогруппа R1a появилась 15 тыс. лет назад в южнорусских степях и пережидала ледниковый период в неком «украинском убежище»? Это приводится, в частности, в той же книге Wells, но откуда это появилось, на основании каких данных — ни слова. Цитирую: «Sometime between 10,000 and 15.000 years ago, a man of European origin was born in presentday Ukraine or southers Russia», Перевод: «Когда-то между 10 и 15 тысяч лет назад мужчина европейского происхождения родился на территории нынешней Украине или южной России». Несколько ранее, в статье 2001 года (Wells et al, Ргос. Natl. Acad. Sci. USA, 98, No. 18, 10244-10249), относительно R1a-M17 он же писал — «М17... with an inferred age of ~15.000 years... originating in Southern Russia/Ukraine», To есть «(мутация) M17 с предполагаемым возрастом примерно 15 тысяч лет... появилась в Южной России или Украине».
Дело вовсе не в том, что статья старая, и что дело давнее, и что все ошибаются. Естественно, в науке ВСЕ ошибаются в той или иной степени. Вопрос не в этом, а в том, откуда данные цифры появились? Анализировали гаплотипы, считали мутации? Естественно, нет, этого они делать не умеют. Просто шли на элементарную подтасовку, основывались не на гаплотипах, не на расчетах, а подгоняли под «общие соображения», выдавая это за науку. Это тоже популяционная генетика.
Именно потому, что анализировать гаплотипы попгенетики не умеют, появились якобы данные, что гаплогруппа R1a имеет «наибольшее разнообразие» на Украине, и там, само собой, ее «прародина». Никаких данных на этот счет опубликовано не было, никакого разнообразия не представлено. Но понятие «украинская гаплогруппа R1a» уже пошло в ход. Wiik в своей статье («Where did European men come from?», J. Genet. Geneal., 2008) повторил слова «украинская гаплогруппа R1a» 37 раз, я не преувеличиваю. На самом деле никакой древности гаплогруппы R1a на Украине нет, возраст практически один — что в Польше, что на Украине, в Белоруссии, в России. Никаких оснований называть гаплогруппу R1a «украинской», «польской» или «русской» нет.
Ho Wikipedia продолжает эти «трактовки» предлагать и сегодня. Вот, например: «The European distribution of Y-chromosome haplogroup R1a has been suggested to have occurred as a result of receding glacial activity allowing males bearing the lineage from the present day territory of Ukraine to migrate and gradually populate central, northern, and western Europe». Перевод: «Было предположено, что распределение гаплогруппы R1a в Европе оказалось результатом ухода ледников, что позволило носителям этой гаплогруппы мигрировать с территории Украины и постепенно заселить Центральную, Северную и Западную Европу». Что было основанием для такого предположения? Судя по описанию, речь идет о временах примерно 12 тыс. лет назад. Откуда на Украине такие датировки для гаплотипов гаплогруппы R1a? Нет таких данных, и не было. Что за работа? Ответ — ссылка на статью Passarino et al, 2002 года в Eur. J. Hum. Genet. Сущие фантазии, ничем не подкрепленные. Популяционная генетика.
С гаплогруппой R1b в Wikipedia не легче. Цитата: «It has been proposed that males from Haplogroup Hg P*(xR1a1) or R1b (Y-DNA) repopulated most of Europe shortly after the Last Glacial Maximum, related to population expansions out of the Franco-Cantabrian region (see also Ahrensburg culture)».
Перевод: «Было предложено, что гаплогруппа R1b перезаселила большую часть Европы вскоре после последнего ледникового максимума, за счет экспансии популяции из Франко-Кантабрианского региона (см. также аренсбургскую культуру)».
К сведению, аренсбургская культура — это верхнепалеолитическая культура на севере Европы, распространенная около 11 тыс. лет назад на восток вплоть до Волги. То, что 11 тыс. лет назад носители R1b могли быть на Средней Волге, продвинувшись с востока — не исключено, но причем здесь Франко-Кантабрианский регион? Ответ, впрочем, ясен — пытались привязать басков, но без анализа гаплотипов. Если бы гаплотипы проанализировали — получили бы 3600 лет назад, но никак не 11 тыс. лет назад. R1b появились в Европе только через 6 тыс. лет. Кто автор сей работы? Ответ: Dupuy, статья 2006 года в журнале Forensic Science International. Чистые фантазии. Вот такая популяционная генетика.
Популяционные генетики Кивисилд и Виллеме активно доказывают, что гаплогруппа R1a появилась в Индии, и оттуда ее носители ушли в Европу. Это они повторяют и в 2011 году, хотя уже известны более «глубинные» субклады гаплогруппы R1a, причем известно, что хронология субкладов идет на понижение (то есть ближе к нашему времени) при переходе их носителей от Европы через Русскую равнину, и далее в Среднюю Азию, Индию, Иран. Не могли эти субклады идти против течения времени из Индии в Европу. Откуда такие «выводы»? Оттуда, что считали мутации «по Животовскому» (см. следующий абзац и далее в настоящей главе), завышая времена в три раза, и получая в Индии 12 тыс. лет вместо правильных 4 тыс. лет для времен жизни общих предков «индоевропейцев». Они, Кивисилд и Виллеме, слышали, что R1a в Европе имеет возраст 4—5 тыс. лет, вот и получается, что это из Индии пришли в Европу, потому что 12 тыс. лет в Индии старше. А все потому, что поленились посчитать сами возраст R1a в Европе, хотя бы для сравнения, тогда получили бы «по Животовскому» в Европе 15 тыс. лет, старше, чем в Индии. А если бы считали в Индии правильно, нашли бы, что там на самом деле несколько популяций R1a, одна — «индоевропейская», возрастом примерно 4000 лет, и другая — древние мигранты с востока, с Гималаев, из Северного Китая, из Южной Сибири. В Индию R1a пришли в основном двумя путями — с востока, примерно 12—10 тыс. лет назад, и с севера, арии, примерно 3500 лет назад. Но популяционные генетики так считать не умеют.
Популяционный генетик Л.А. Животовский нанес трудно поправимый вред своей науке, введя совершенно искусственные «популяционные скорости мутаций». Они имеют совершенно узкое и практически нереальное применение, базируются на нереальных предположениях и допущениях, и фактически завышают времена жизни общих предков популяции в 2,5—3 раза (см. далее в настоящей главе). Более того, чтобы «обосновать», что эти ложные «популяционные скорости мутаций» применимы к реальным системам, Л.А. Животовский произвел откровенную подгонку данных, меняя состав изучаемых серий гаплотипов, меняя полученные «скорости мутаций» и подтягивая цифры к желаемым, и приводя названия популяций, которые он якобы анализировал, а на самом деле НЕ анализировал. Это все было объявлено как доказательство применимости «популяционной скорости мутации». Подробное рассмотрение этих манипуляций — в последующем разделе настоящей главы, а также в работах «Еще раз о «популяционной скорости мутаций» Л. Животовского, или как рождаются басни» (Клёсов, 2009), и в статье в журнале Human Genetics (Klyosov, 2009).
Понятно, что все могут ошибаться, и от ошибок никто не застрахован, в том числе и в науке. Собственно, наука и развивается так, что при взгляде назад можно увидеть цепь ошибок, которые были вызваны отсутствием важных данных, и соответственно неверными, неполными, и просто ошибочными интерпретациями. В науке это — нормальное дело. Но я здесь говорю про другие ошибки, которыми изобилует популяционная генетика, а именно отсутствием умения и желания анализировать ИМЕЮЩИЕСЯ данные, рассматривать альтернативы, отсутствием способности рассматривать имеющиеся данные в совокупности, сравнивая их и сопоставляя. Примеров — масса. Собственно, практически каждая статья в области «популяционной генетики человека», выходящая в настоящее время в «академических» журналах, включая Доклады Академии наук США, Доклады Британского королевского общества, не говоря о специализированных журналах в области генетики человека и популяционной генетики, представляет собой нагромождение нелепиц. Это — бич современной популяционной генетики.
Причина, на мой взгляд — отсутствие разумной, критической, добротной современной школы в популяционной генетике человека, в той ее части, которая занимается изучением истории человечества на основе гаплогрупп и гаплотипов Y-хромосомы и мтДНК. Соответствующие примеры нелепиц мы разбираем почти в каждом выпуске «Вестника Академии ДНК-генеалогии», не для того, чтобы научить попгенетиков, это бесполезно, они уже необучаемы, но для того, чтобы на их примерах учить молодых ученых, как не надо анализировать данные, и как надо это делать.
Работа по переделке материала и выводов статей попгенетических авторов практически бесконечна, потому что, повторяю, они безграмотны, в них сильно заметно отсутствие нормальной научной школы. Поэтому дело не столько в популяционной генетике, сколько в популяционных генетиках.
Но, как говорил Козьма Прутков, порой и терпентин на что-нибудь полезен. Работа с этими статьями шлифует методологию ДНК-генеалогии, позволяет получать новые результаты и выводы, по ассоциации привлекаются новые материалы, которые опять же анализируются. Далее, в их статьях приводятся фактические данные, гаплотипы и гаплогруппы, из которых попгенетики делают, как правило, совершенно примитивные и неверные выводы, но сам фактический материал можно использовать. Ссылки на эти фактические материалы я всегда даю. Так что как собиратели материала, как техники-лаборанты попгенетики приносят несомненную пользу. Сами же статьи их лучше не читать, а если читать, то понимая, что их интерпретации, и вообще раздел «Обсуждение результатов» в каждой статье надо пересматривать заново. Тогда все в порядке.
После такого введения рассмотрим несколько примеров анализа данных попгенетиками, то, какие выводы они делают, и то, какие выводы на самом деле стоило было бы сделать, если вместо «популяционной генетики» приложить подходы ДНК-генеалогии.
Статья довольно старая, 2004 года (Kasperaviciute et al, 2004). Но она опубликована в заслуженном журнале Annals of Human Genetics, в ее авторах — известный популяционный генетик М. Stoneking, и сама статья — о гаплотипах литовцев, близкая нам популяция.
Статья, на мой взгляд, не особенно интересная, и единственное, что в ней привлекает внимание, это то, что она ставит задачу показать происхождение современной популяции литовцев. Собственно, это и должна была быть главная задача статьи, это же вынесено в резюме статьи: to address questions about the origin... of the present day population. Посмотрим, к какому выводу пришли авторы и как они это сделали.
Ответ, собственно, уже дан в самом резюме статьи: ап analysis of molecular variance confirmed the homogeneity of the Lithuanian population. To есть анализ некого «молекулярного разнообразия» подтвердил гомогенность современной популяции литовцев. Вот это уже интересно. Мы привыкли к тому, что в любой популяции есть свои ДНК-генеалогические ветви, что любая популяция состоит из разных гаплогрупп, то есть первичных родов, что любая популяция в этом отношении гетерогенна, просто одни популяции более гетерогенны, чем другие.
Поэтому крайне странно, что литовская популяция провозглашается «гомогенной». На самом деле по данным самих же авторов среди 196 изучаемых гаплотипов мужчин-литовцев к гаплогруппе R1a относилось 88 человек (45%), к N1c1 — 72 человека (37%), к сводной гаплогруппе BR*(xDE, JR) — это, видимо, в основном гаплогруппа I — 20 человек (10%), к гаплогруппе P*(xR1a) — это, видимо, в основном гаплогруппа R1b — 10 человек (5%), плюс минорные гаплогруппы DE (5 человек, или 2,6%) и K*(xN1c1, Р) — 1 человек. Какая же здесь «гомогенность», когда доминирующая гаплогруппа (R1a) охватывает менее половины мужского населения?
Как водится у попгенетиков, авторы посчитали «разнообразие» (diversity) — некий полуколичественный параметр — всех гаплогрупп, и нашли, что он равен 0,653±0,020, У эстонцев он оказался равен 0,741±0,012, у латышей 0,667±0,021, у русских 0,712±0,031. Иначе говоря, вариации по гаплогруппам у литовцев были несколько меньшими по сравнению с другими перечисленными популяциями. Тем не менее, хороша «гомогенность».
Оказывается, под «гомогенностью» авторы подразумевают, что все шесть основных этнолингвистических групп литовского населения неоднородны одинаково, никакая при этом не выделяется в ту или иную сторону. Они гомогенны в своей неоднородности. И это понятно, потому что по сообщению самих же авторов они, эти этнолингвистические группы, образовались в ходе последнего тысячелетия, когда гаплогруппы были уже перемешаны. Так что вопрос о «происхождении современной литовской популяции» остался пока без ответа, перейдя в другую плоскость — что эта популяция была уже перемешана тысячу лет назад. О происхождении это, конечно, ничего не говорит.
Далее авторы занялись гаплотипами в 9-маркерном формате, и началась типичная попгенетическая фантастика. Авторы отметили, что среди 196 гаплотипов 123 являются разными. Стало быть, 73 гаплотипа повторяются, причем что это знание дает — авторы, конечно, не представляют и об этом не пишут. Ответ — не дает ничего, кроме пустых цифр. Авторы нашли, например, что среди 88 гаплотипов гаплогруппы R1a у литовцев 56 гаплотипов были «уникальными» («дистинктными»), и семь из них были «наиболее одинаковыми»,
(переписаны здесь в формате FTDNA для 12-маркерных гаплотипов). Эти семь, как пишут авторы, составляют «3,6% от всех Y-хромосом в литовцах» (то есть 7 из 196), не понимая, что фраза эта лишена какого-то либо определенного смысла и никому в таком виде не нужна. Но «так принято», потому что разделить одно на другое несложно, а потом записать и отправить в статью, а зачем — это уже не важно.
По секрету — из этой информации уже можно определить возраст гаплогрупп по популяциям, но попгенетики об этом не имеют ни малейшего понятия. Как определить — будет пояснено ниже.
То же самое относится и к гаплотипам гаплогруппы N1c1 у литовцев, для которых авторы нашли, что из 72 человек четверть (то есть 18 человек) имеют идентичные гаплотипы
Авторы опять записали бессмысленную «информацию», что это составляет 22,4% от всех литовских Y-хромосом (разных гаплогрупп).
То, что написано в обсуждаемой статье популяционных генетиков, причем написано совершенно безосновательно: «Alternatively, the Baltic tribes from which modern Lithuanians originated may have been genetically homogeneous», не соответствовало действительности. Перевод: «Как вариант, балтийские племена, из которых образовались современные литовцы, были генетически гомогенными». Мы уже знаем, что под «генетически» авторы понимают гаплотипы и гаплогруппы. На самом деле у авторов нет никаких конкретных данных о древних балтийских племенах, и данные статьи об этом совершенно не говорят. Датировки, полученные в статье, неверны (см. ниже), интерпретации — сплошное недоразумение. Популяционная генетика.
Вместо обоснованных и информативных расчетов авторы обсуждаемой статьи зафиксировали, что «gene diversity based on 9 STR loci was 0,985±0,004». Для начала, никаких gene, то есть «генов», в этих 9-маркерных гаплотипах нет. Это просто небрежный, вульгарный сленг. Речь идет о негенных, нерекомбинантных фрагментах ДНК. Далее, разнообразие, по понятиям авторов, высокое, и не мудрено, для разных-то гаплогрупп. А вот в пределах гаплогруппы N1c1 у литовцев разнообразие по данным авторов было минимальным — 0,915±0,023. Опять остается загадкой, зачем все это считать, потому что ни для чего дальше эти цифры в статье не используются, не применяются и не анализируются. В рамках ДНК-генеалогии ясно, что такие цифры связаны (прямо или косвенно) с возрастом популяции, но поскольку здесь смешаны в кучу серии гаплотипов из разных ветвей, то возраст из этих цифр «разнообразия» не посчитать, и никому они, эти цифры, не нужны. Просто «так принято», совершенно бездумно. Ниже мы покажем, как это на самом деле надо делать. А пока заметим, что по данным авторов для литовцев в пределах гаплогруппы R1a это «разнообразие» выше — 0,984±0,05, и понятно, почему: гаплогруппа R1a старше по сравнению с N1c1 в литовской популяции, как будет показано ниже в наших расчетах. Им в данных популяциях 4375 и 2400 лет, соответственно (см, ниже).
Занятно, что авторы, не разбираясь в скоростях мутаций, практически вслепую применили как скорости мутаций Kayser et al (2000), основанные на парах отец-сын (полученные при совершенно недостаточной статистике, и, видимо, приводящие к заниженным датировкам), так и «популяционные скорости мутаций» Л. Животовского, которые приводят к завышениям датировок примерно в три раза. Так и получилось — первые дали 900 лет до общего предка литовцев гаплогруппы N1c1, вторые — 7600 лет, в 3,2 раза выше чем по более корректным расчетам (см. ниже). Для гаплогруппы R1a у литовцев соответствующие датировки получились 1100 и 7800 лет, соответственно, при более правильной величине 4375 лет до общего предка (см. ниже). Особенно поразительно, что авторы включили в свои расчеты и время жизни общего («усредненного»!) предка популяции по всем гаплогруппам одновременно (!), что вообще не имеет никакого физического смысла. У них получилось 1000 и 7000 лет «по Кайзеру и по Животовскому», соответственно. Здесь комментариев просто нет.
Верные своим принципам, популяционные генетики тут же подгоняют полученные цифры к устоявшимся данным, полученным представителями других наук, в данном случае, в археологии и антропологии. Авторы пишут, что полученная величина в 1000 лет («по Кайзеру») согласуется с временем развития сельского хозяйства на территории современной Литвы. На самом деле то, как эта цифра была авторами получена, является ошибкой. Сельское хозяйство здесь не при чем. Сюда же относятся фантазии о «элитном доминировании», тоже любимом предмете популяционных генетиков, который вставляется к месту и ни к месту, как в данном случае, поскольку расчеты датировок неверные. Сюда же относится невразумительная фраза «the observed pattern of Y chromosome diversity in Lithuanians may be explained by a population bottleneck associated with Indo-European contact», помещенная в резюме. Какая «наблюдаемая картина разнообразия», о чем здесь речь — остается неизвестным, как и при чем здесь «бутылочное горлышко популяции», у кого, и откуда оно появилось — тоже остается без ответа, как и то, что за «индоевропейцы» здесь упоминаются, видимо, древние носители гаплогруппы R1a. Но почему они создали «бутылочное горлышко», когда носители гаплогруппы N1c1 просто пришли в Прибалтику позже — загадка сия велика есть.
Как видно, материал статьи совершенно бессвязный, вопрос о происхождении литовцев остался, естественно, без ответа, датировки неверные, формальные цифры «разнообразия» остались без интерпретации, «элитное доминирование» и «бутылочные горлышки популяции» неясно зачем вообще введены, к тому же диапазон датировок от 900 лет до 7600 лет для одних и тех же событий (в данном случае для общего предка гаплогруппы N1c1 у литовцев) делает задачи статьи лишенными смысла. Популяционная генетика.
Для сравнения приведем здесь расчеты по литовским мужским популяциям с помощью методов ДНК-генеалогии. Для начала — используем данные самих же авторов рассматриваемой статьи, и применим методы расчетов, авторам, видимо, незнакомые.
Итак, авторы нашли, что среди 88 гаплотипов гаплогруппы R1a у литовцев семь были идентичными друг другу, и имели вид
В понятиях ДНК-генеалогии это — базовые гаплотипы. Для данного 9-маркерного формата гаплотипов константа скорости мутации равна 0,017 мутаций на гаплотип на условное поколение в 25 лет, что дает [ln(88/7)]/0,017=149→175 поколений, то есть примерно 4375 лет до общего предка литовских мужчин гаплогруппы R1a в данной популяции, что в пределах погрешности совпадает с возрастом общего предка гаплотипов R1a на Русской равнине. И гаплотип у общего предка тот же самый, что в усеченной форме записан выше:
Что касается «гомогенности», то следующий рисунок показывает, что никакой гомогенности в литовской выборке гаплотипов R1a не наблюдается. Литовские гаплотипы расходятся по разным ветвям, с общими предками, которые жили (определено по значительно более массивным выборкам):
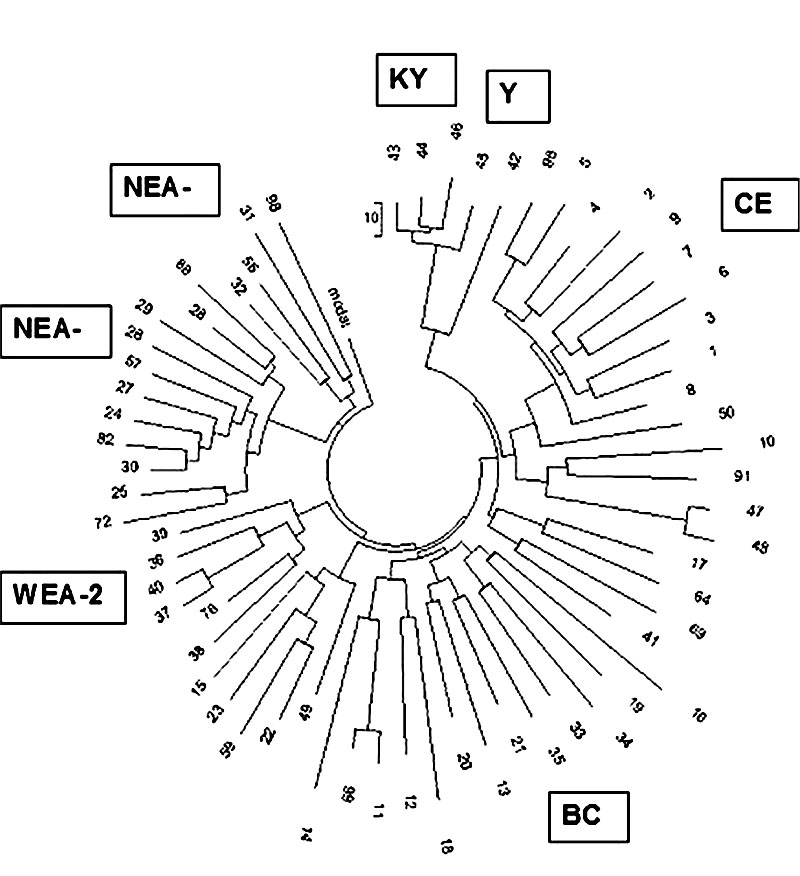
— Центрально-европейская ветвь — 3425 лет назад (она в свою очередь расходится на две подветви с возрастом 3000±400 лет каждая); у литовцев там 94 мутации от 67-маркерного базового гаплотипа ветви, что составляет 94/9/0,12=87→96 поколений, то есть 2400±350 лет до общего предка.
— Балто-карпатскую ветвь с общим предком 4000±500 лет назад, и двумя подветвями с общими предками 2700±300 и 2500±300 лет назад; литовские гаплотипы принадлежат всем трем подветвям.
— Западную евразийскую ветвь-2 возрастом 2600±300 лет; пять литовских гаплотипов отклоняются суммарно на 51 мутацию от базового гаплотипа, что дает 51/5/0,12=85→93 поколения, то есть 2325±400 лет до общего предка, в хорошем соответствии с возрастом всей ветви.
— Северную евразийскую ветвь возрастом 2200±250 лет; 15 литовских гаплотипов этой ветви расходятся на две подветви, NEA-1 и NEA-2.
— Еще есть серия южно-азиатской ветви, с гаплотипами, похожими на киргизские и общим предком примерно 1050 лет назад. Это, скорее всего, литовские татары.
Эти ветви все в совокупности идут от общего предка Русской равнины, который жил примерно 4800 лет назад.
При рассмотрении 72 гаплотипов гаплогруппы N1c1 у литовцев авторы обсуждаемой статьи нашли, что из них четверть (то есть 18 человек) имеют идентичные гаплотипы
Считать «разнообразие» — здесь давно устаревшее понятие. Вместо этого запишем [1п(72/18)]/0,017=82→90 поколений, то есть примерно 2250 лет до общего предка литовских мужчин гаплогруппы N1c1 в данной популяции. Это — конец прошлой эры. Посмотрим, что дают более аккуратные расчеты по большим выборкам более протяженных гаплотипов. Базовый гаплотип 20 литовцев гаплогруппы N1c1- M178+L708+L550+ из базы данных FTDNA имеет вид
и все 20 37-маркерных гаплотипа имеют 152 мутаций от приведенного базового гаплотипа. Это дает 152/20/0,09=84→92 поколений, то есть 2300±300 лет до общего предка. Совпадение практически абсолютное с расчетом выше по данным обсуждаемой статьи. В целом среди N1c1 преобладает DYS19=14, у литовцев — 15. В пределах гаплогруппы N1c1 литовцы образуют южно-балтийскую ветвь с характерной четверкой DYS464 14-14-15-15 и DYS459 9-9, в отличие от финской ветви с 13-13-14-14 и 10-10, соответственно. Правда, и там литовские гаплотипы расходятся на несколько ДНК-генеалогических ветвей с общими предками в середине 1 тыс. н.э., примерно 1500 лет назад.
Но это — суммарные расчеты, без учета того, что дерево гаплотипов литовцев гаплогруппы N1c1 на самом деле расходится на ветви, и у каждой ветви — свой возраст, свой общий предок. Расчеты показывают, что все эти ветви образованы в середине 1 тыс. н.э. или позже. Посмотрим, как это делается.
На дереве отчетливо видны две обширные ветви справа и слева, которые в свою очередь состоят суммарно из семи подветвей.
Ветвь 1 из шести гаплотипов имеет базовый гаплотип
В ветви — 30 мутаций, что дает 30/6/0,09=56→60 поколений, то есть 1500±310 лет до общего предка ветви. Это — середина 1 тыс. н.э.. Как раз время подъема сельского хозяйства в регионе, согласно цитируемым авторами рассматриваемой статьи археологических данных. Только это уже не «между 1000 и 7000 лет», а вполне корректно рассчитанные данные.
Ветвь 2 рядом — это не дочерняя ветвь, она происходит от предка примерно 1050 лет назад, и имеет вместе с первой ветвью общего предка примерно 2200 лет назад. Две подветви 3 и 4 легко объединяются, так как они одинаковые по размеру, и «вес» их одинаков. Эти шесть гаплотипов имеют базовый гаплотип
В этой ветви тоже 30 мутаций, что тоже определяет возраст ветви в 1500 лет назад.
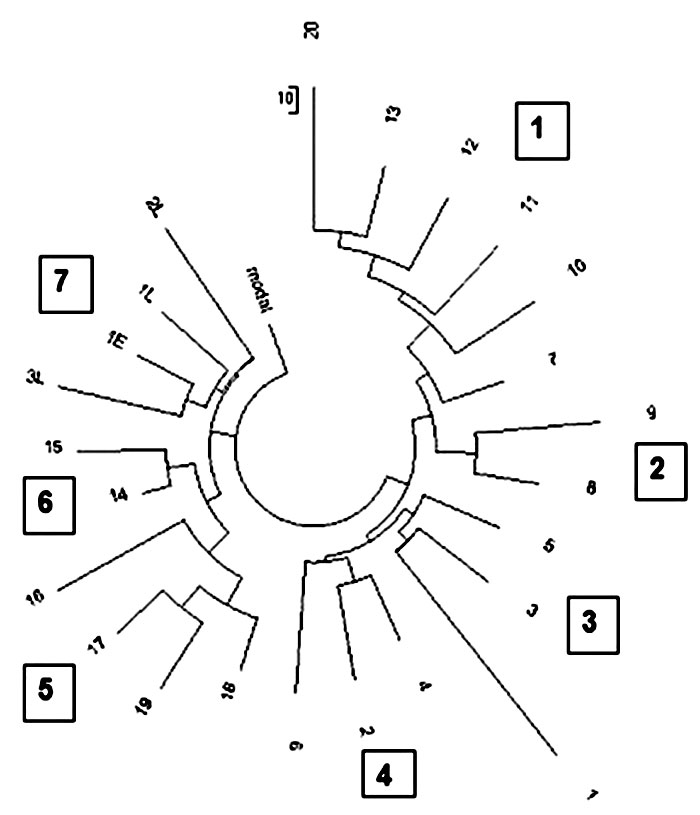
Между ветвями 1 и 3-4 — 6 мутаций, что разводит их общих предков на 6/0,09=67→72 поколений, то есть на 1800 лет, и помещает их общего предка на (1800+1500+1500)/2=2400 лет назад.
Ветвь 5 имеет базовый гаплотип →
и 14 мутаций ветви от этого базового гаплотипа помещают общего предка ветви на 1025±280 лет назад.
Малая ветвь рядом (6) имеет недавнего предка, всего 425 лет назад. Но она отстоит от ветви 5 на 6,5 мутаций, то есть на 1950 лет, что помещает общего предка ветви 5—6 примерно на 1700 лет назад.
Наконец, сводная ветвь латышей и эстонца (7) имеет возраст 1250 лет. Но она заметно отличается по своему базовому гаплотипу
от остальных ветвей. Так, от соседней ветви 5 она отличается на 8 мутаций, что помещает их общего предка на 2400 лет назад.
Как мы видим, примерно 2200—2400 лет назад жил общий предок южно-балтийской ветви гаплотипов группы N1c1, который принципиально отличался от финской ветви по ряду маркеров в базовом гаплотипе, как описано выше. Подавляющее большинство литовских, латышских и эстонских гаплотипов группы N1c1 относятся именно к южно-балтийской ветви, с четверкой 14-14-15-15 (DYS464) и парой 9-9 (DYS459). Насколько эти различия с финской группой выражены и воспроизводимы, видно из дерева гаплотипов. Надписи «литовцы» и «финны» показывают, в какой ветви преобладающе оказываются эти этнические группы. Это дерево «четверок» гаплотипов напоминает то, что приведено выше, в главе 14, для «шестерок» гаплотипов. «Шестерки» более точные, чем «четверки», но и здесь разрешение прекрасное.
Гаплогруппы I2 у литовцев практически нет, как и вообще в Прибалтике. По информации базы данных проекта FTDNA, среди 594 гаплотипов гаплогруппы I2 имеются только один литовский, один латышский и ни одного эстонского гаплотипа.
Аналогично, по данным другого проекта, среди 3400 гаплотипов есть только четыре прибалтийских, эстонский и три литовских гаплотипа.
Эти три литовских гаплотипа гаплогруппы И имеют следующий базовый гаплотип в 67-маркерном формате:
Во всех 67 маркерах они имеют 25 мутаций, что дает 25/3/0,12=1850±410 лет до общего предка. Это явно заниженная датировка, потому что гаплотипов всего три. Ниже показано, что при увеличении выборки до 10 гаплотипов датировка возрастает до 3000±430 лет до общего предка.
Если к этим трем гаплотипам литовцев добавить гаплотипы эстонца, украинца, двух белорусов и трех русских той же гаплогруппы, то полученные 10 гаплотипов имеют тот же базовый гаплотип, но с датировкой общего предка 2175±340 лет назад.
Надо, впрочем, принять во внимание, что эти 10 гаплотипов принадлежат разным субкладам (см. схему ниже) — один литовец субклада Ild-L22 (как и эстонец, все трое русских и украинец), один литовец и два белоруса — субклада I1b-M227, и один литовец — общего субклада I1, что, впрочем, означает, что субклады для него не определяли. Поэтому в реальности общий предок всех трех литовских гаплотипов может быть древнее.

В базе данных Ysearch обнаружились 10 литовских гаплотипов, и в составе 74 восточноевропейских гаплотипов они приведены на дереве 25-маркерных гаплотипов:
и 14 мутаций ветви от этого базового гаплотипа помещают общего предка ветви на 1025±280 лет назад.
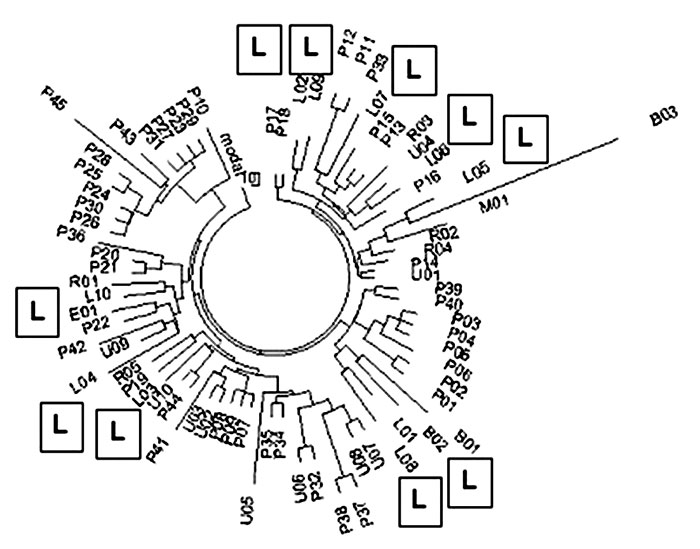
Видно, что литовские гаплотипы не занимают какого-либо определенного места, не формируют свою ветвь, как и любые другие восточно-европейские гаплотипы. Все 74 гаплотипа дерева имеют базовый гаплотип (две отличающиеся мутации от предыдущего базового гаплотипа отмечены)
и суммарно отстоят от него на 386 мутаций. Это дает 386/74/0,046=113→128 поколений, или 3200±360 лет до общего предка.
Еще 10 гаплотипов приведены и на литовском сайте. Базовый гаплотип там следующий:
Он практически такой же, что приведен выше для серии гаплотипов с Проекта I1 FTDNA. В 37-маркерном формате все 10 гаплотипов имеют 96 мутаций, что дает 96/10/0,09= 107→120 поколений, то есть 3000±430 лет до общего предка. Это — в пределах погрешности с величиной, данной выше.
Надо опять принять во внимание, что эти 10 гаплотипов принадлежат разным субкладам — один литовец к субкладу I1d1, и один — к субкладу I1b-M227.
Как видно, самая древняя гаплогруппа у литовцев — R1a (середина 3 тыс. до н.э.), и она же самая распространенная. Следующей по древности идет гаплогруппа I1 (конец 2 тыс. до н.э. — начало 1 тыс. до н.э.), и далее — гаплогруппа N1c1, которая вошла в литовскую популяцию только в середине 1 тыс. н.э.
Итак, рассмотрение литовских гаплотипов разных гаплогрупп показывает, что общий предок гаплогруппы R1a литовцев жил примерно 4600 лет назад, в середине 3 тыс. до н.э., и прибыл на Балтику в составе разных субкладов, почти все из них образовались в середине 1 тыс. до н.э. (центрально-европейский 2400±350 лет назад, балто-карпатский 2500 и 2700 лет назад, западный евразийский 2600±300 лет назад, северный евразийский 2200±250 лет назад). Носители этих субкладов говорили, как показывает лингвистика, на диалектах древнего арийского языка, что и образовало балтскую языковую группу, или балто-славянскую, в зависимости от того, как называть и чему отдавать предпочтение.
Как отмечает И.Л. Рожанский (2011), по меньшей мере три линии миграции восточных славян в Литву, хотя и крайне слабо представленных там, можно связать с миграциями из карпатского (или дунайско-карпатского) региона. Это обе ветви субклада М458 и восточная карпатская (она же западно-евразийская-2) ветвь субклада Z280, Все они имеют почти один и тот же возраст, около 2700 лет.
Примерно одновременно с ними на Балтику стали прибывать носители гаплогруппы I1, значительно меньшие по численности. Их источник — Европа, где гаплогруппа I1 прошла «бутылочное горлышко» популяции примерно 3400 лет назад, и Восточная Европа с общим предком 3200±360 лет назад, примерно то же, что и у литовских гаплотипов. Это, видимо, и были древние балты, во всяком случае, нынешние литовцы — их прямые потомки. Те, кто жили на территории современной Литвы раньше, если таковые были, видимо, не оставили потомства, которое бы дожило да нашего времени.
Заметно позже, к середине 1 тыс., в Прибалтику стали прибывать носители гаплогруппы N1c1, ее южно-балтийской вегви, отделясь от финской ветви, которая прошла и расселилась севернее. Общий предок обеих ветвей жил на Урале, или находился на миграционном пути между Уралом или Поволжьем и Балтикой примерно 3200 лет назад, и эта миграция расщепилась на несколько ДНК-генеалогических линии, все примерно одинакового «возраста», все датируются серединой 1 тыс. н.э. Случайное это совпадение или нет, но в те же времена в Европе происходило становление языкового славянства, и в те же времена вдоль Прибалтики формировались прибалтийские племена, а также викинги. Одна линия сформировала угро-финскую ветвь, состоящую в значительной степени из финнов, но с добавлениями части русских, норвежцев, шведов; другая линия дала потомков, ныне живущих в основном в России, в прибалтийских странах — Эстонии, Литве, Латвии, а также Польше, и их соседей, откуда гаплотипы южно-балтийской группы попали с потомками вплоть до Атлантики и Мексики. Характерными признаками гаплотипов угро-финской группы являются DYD459=10-10, DYS464=13-13-14-14, негативный снип L550-. Характерными признаками гаплотипов южно-балтийской группы являются DYD459=9-9, DYS464=14-14-15-15, позитивный снип L550+. Сами литовцы впервые упоминаются в исторических документах в начале XI в., то есть всего несколько столетий после прибытия носителей гаплогруппы N1c1.
Неолитические обитатели Балтики начиная с 6500 лет назад в гаплотипах современных литовцев не просматриваются. Данные об «интенсивном культурном развитии в Литве, включая керамику», возможно, и были, но, похоже, потомков тех людей в Прибалтике не осталось. Что касается балтов «в 5-м тысячелетии назад», то это могли быть носители гаплогруппы R1a из Европы и с Русской равнины. Авторы справедливо пишут, что «археологические данные показывают, что финно-угорское влияние на литовскую популяцию было минимальным». Правда, авторы это ничем не подтверждают, и просто цитируют данные археологии. ДНК-генеалогия это полностью подтверждает. Носители гаплогруппы N1c1 прибыли на Балтику уже относительно поздно, в середине 1 тыс. н.э., и, самое главное, это не были финно-угры по гаплотипам. Те ушли севернее, а южнее, в литовской Прибалтике, были южные славяне южно-балтийского субклада гаплогруппы N1c1.
Статья (Морозова и др.) относительно недавняя, опубликована в декабре 2011 года. Авторы — группа сотрудников Института общей генетики РАН. Удивительно, что среди авторов нет Л.А. Животовского из того же института, но такую независимость и самостоятельность можно только приветствовать.
Изначально к статье симпатия, поскольку любой толковый материал про этническую историю русских можно только приветствовать. Что можно ожидать от такого материала? Давайте начнем скорее с того, что уже известно, в целом или в частности. Известно, что мужской состав этнических русских в основном определяют три гаплогруппы — R1a, N1c1, и I (11+12), на которые приходится суммарно примерно 84%. Это, условно говоря, восточные славяне (доля в Европейской части России в среднем 48%, но до 63% в южных областях России, и соответственно меньше на севере, от Пскова и севернее), южные балты, на которых приходится в среднем 14% по Европейской России, и, условно говоря, общеевропейские гаплогруппы I1 и I2, с их 6,5% и 15%, соответственно. Последние две гаплогруппы называют то «палеоевропейскими», то «древнерусскими», то «балтийскими», «скандинавскими» (I1), и «южнославянскими» или «балканскими» (I2). Ни одно из этих названий не является абсолютным, и полностью зависит от контекста, в который эти названия помещают.
Например, «восточнославянской» гаплогруппа R1a является только на территории России, и то только на основании лингвистических категорий. Название «южные балты» для гаплогруппы N1c1 применимо для тех, кто живет в России, Литве, Латвии, Эстонии, Польше, потому что они действительно принадлежат в основном южно-балтийской ветви гаплогруппы N1c1, со снипом L550 и характерными аллелями в гаплотипах (например, двойкой 9-9 и четверкой 14-14-15-15 в DYS459 и DYS464, соответственно). Та же гаплогруппа N1c1, но среди этнических финнов (то есть тех, кто считают себя финнами, говорят на финском языке и поколениями живут в Финляндии) южно-балтийской уже никак не являются, имеют негативный снип L550, двойку 10-10 и четверку 13-13-14-14, соответственно.
Что касается гаплогрупп I1 и I2, то в России их совсем мало по сравнению с Британскими островами и вообще Европой. Поэтому по сути это «общеевропейские» гаплогруппы, хотя есть основания считать, что гаплогруппа I могла возникнуть на Русской равнине более 40 тыс. лет назад, и затем мигрировать на запад, в Европу. Возможно, там и разошлась на субклады I1 и I2, причем первый явно прошел «бутылочное горлышко» популяции примерно 3500 лет назад, в середине 2 тыс. до н.э., и в небольших количествах вернулся на Русскую равнину примерно 3200 лет назад. В применении в России их можно считать условно «южнорусскими», потому что, например, в Прибалтике их практически нет. Из тысяч гаплотипов проектов I1 и I2 только десяток прибалтийских I1, причем они есть только в литовском проекте, в котором 125 гаплотипов гаплогруппы N1c1 и 60 гаплотипов R1a. В крупном проекте I2 среди 594 гаплотипов гаплогруппы I2 имеются только один литовский, один латышский и ни одного эстонского гаплотипа. Там же — 9 русских, 2 украинских и 6 белорусских гаплотипов. В еще более крупном проекте I1 среди 3400 гаплотипов есть только один эстонский и 3 литовских гаплотипа, но 12 русских, 9 украинских и 3 белорусских. Эти данные можно, конечно, интерпретировать поразному, но суть в том, что гаплотипов I1 и I2 на севере мало, и они смещены к югу. Назвать гаплогруппу II «прибалтийской» в контексте Русской равнины было бы неуместно.
Что касается женских мтДНК, то в целом известно, что преобладающей гаплогруппой в Европейской части России является гаплогруппа Н (примерно 43%), а также гаплогруппы U5 (11%), Т2 (9%),J (примерно 8%), остальные — в меньшей степени.
В отношении «этнической истории» России на основании мтДНК трудно представить что-либо новое или необычное, что такое исследование может дать, исходя из общих представлений. ДНК-генеалогию там особо не вычислить, потому что одна мутация в мтДНК происходит раз в несколько тысяч лет. С такими скоростями мутаций «этническую историю русских» изучать трудно, если под «русскими» не считать тех, кто жили на Русской равнине 5—10 тыс. лет назад. Более того, для всех трех основных мужских гаплогрупп их доля по областям Российской Федерации почти полностью неупорядочена и перемешана. Есть только общая тенденция роста доли гаплогруппы N1c1 на север (начиная с широты Пскова) и роста доли R1a, напротив, на юг. По общим соображениям, мтДНК тоже должны быть там, в русских областях, перемешаны. Сколько тысячелетий прошло с их образования и множественных миграций, притом что женщины обычно переходили в замужество на другие территории. Но это — общие соображения, которые порой не подтверждаются объективными научными данными.
Посмотрим, что нашли в этом отношении авторы. Открываем статью с надеждами. Читаем абстракт. Там обычно в концентрированном виде то, что НАШЛИ, а не то, что ДЕЛАЛИ. По крайней мере, так должно быть.
Итак, если отжать воду, а именно что «делали», то имеем:
1. Подтверждено, что средневековые славяне имели племенную структуру.
2. Показано, что «разнообразие» среди русских начинается во второй половине 1 тыс. н.э., «времени колонизации Восточно-Европейской равнины славянскими племенами».
Как, и это все? Кто бы сомневался в этих основных выводах, тем более что это все взято из учебников истории для средней школы.
(Более того, как показано ниже, это из данных авторов совершенно не следует. Но наберемся терпения. Перед нами — интересный и поучительный случай).
Ну ладно, уже ясно, что ничего нового не нашли. Или в известном ключе популяционной генетики новые выводы сделать просто побоялись, и подогнали к тому, что уже известно. Хорошая наука, удобная, неконфликтная.
Правда, в абстракте есть еще загадочная фраза, что «картина разнообразия объяснена как влиянием нативных популяций Восточно-Европейской равнины, так и генетическими различиями среди ранних славян». Ну так почему не написали, что это дало? Что значит «объяснена»? Вывод-то какой сделан? Абстракт именно для того, чтобы вывод написать, а не поделиться, типа, что «мы работали».
Вот это и есть отсутствие грамотной школы в популяционной генетике. Никто не научил, что в абстракт выносят то, что найдено, как продвинута наука в результате исследования, а не то, что делалось. Типа «мы собирали грибы». А где грибы-то? «Да мы собирали».
Ладно, если даже ничего нового не нашли, но грамотно показали, что средневековые славяне делились на племена, и что общие предки славян жили во второй половине 1 тыс., уже хорошо для становления науки, пусть будет грамотным упражнением. Но уже закрадываются сомнения, потому что по Y-хромосомным гаплотипам известно, что ветви славянских (или праславянских) племен ведут свое начало в основном из начала-середины 1 тыс. до н.э., а общие предки этих племен жили на Русской равнине примерно 4900 лет назад. В те же времена или чуть позже предки нынешних славян продвинулись в Прибалтику, на территории современных Литвы и Латвии. Примерно 4000 лет назад предки нынешних славян прошли до Южного Урала, основали андроновскую и синташтинскую археологические культуры, и примерно 3500 лет назад предки нынешних славян, точнее, братья наших предков, ушли в Индию, Иран, Анатолию, Саудовскую Аравию. Ушли, естественно, с Русской равнине, не по воздуху летели из Европы.
Это была долгая миграция, с образованием племен, ветвей гаплотипов, образованием новых субкладов гаплогруппы R1a. Хотя на север Русской равнины, в Южную Прибалтику носители южно-балтийской ветви гаплогруппы N1c1 прибыли действительно в середине 1 тыс. н.э. Но перед этим там уже были носители гаплогруппы R1a (см. выше), что и сформировало балтов и привело к образованию балто-славянской языковой группы. Но явно неверно ограничивать наших славянских, или русских предков только «второй половиной 1 тыс. н.э.», это только племена гаплогруппы N1c1, ставшие южно-балтийскими. Может, авторы рассматриваемой статьи только их изучали? Смотрим дальше, саму статью.
Набор рассматриваемых мтДНК впечатляет. Помимо 953 образцов из России (образцы из 10 областей собирали сами, плюс пять областей добавили из литературы, всего получив образцы из 14 областей России, одна область перекрывалась по своим и литературным данным). Еще добавили массу литературных данных по мтДНК, что позволило получить репрезентацию по мтДНК по шести языковым группам — славянской, балтской, финно-угорской, тюркской, германской, и иранской (последние — осетины и персы). Естественно, языковые группы здесь ни при чем, в ДНК они не отражены. Речь идет о территориях преимущественного расселения носителей этих языков, причем в наше время. В древности эти (языковые) территории часто были, естественно, другие. Неясно, почему авторы так напирают на языки. Это у них, видимо, такой сленг, не очень удачный.
Для начала — сразу относительно мелкое замечание. Авторы много места уделяют описаниям статистических расчетов, как и положено популяционным генетикам, но на практике вся эта теория отбрасывается. Например, авторы пишут, что средняя частота восточно-евразийских гаплогрупп среди восточноевропейских российских областей составляет 2,44%, а среди поляков, например, только 1,8% (или максимум 1,8%). Это понятно — где поляки и где Восточная Азия. Но на самом деле здесь не 2,44%, да еще с точностью до сотых долей процента. Цифры по всему полю прыгают, и, например, гаплогруппа A почему-то подскакивает до 3,23% у смолян (на самом деле там 2 человека из 62 смолян, какие уж там «3,23% до сотых долей процента», статистическая погрешность при наличии всего двух человек как минимум плюс-минус 70%, и то это для одной сигма, для двух сигма погрешность вдвое выше, что вообще почти теряет смысл). У рязанцев гаплогруппу A имеет один человек из 60, и еще у новгородцев — один из 114 человек. У остальных 11 областей— ни одного. Чего уж там усреднять, да еще до сотых долей процента... Вот оно, отсутствие грамотной научной школы. Короче, восточноазиатских гаплогрупп в российских областях по представленным данным 2.45±1,50%, а у поляков — 1,8%, и тоже с погрешностью. Так что это одни и те же цифры, у русских и поляков, в пределах погрешности.
Вот это и огорчает, что авторы не понимают, или пренебрежительно относятся к тому, как надо представлять данные. Это тоже результат отсутствия у них хорошей научной школы. Это и сыграло с ними фатальную шутку, как станет ясно ниже. Надо же понимать, что складывая, вычитая и деля цифры статистической природы, надо особенно внимательно представлять интервалы погрешности, доверительные интервалы. Нельзя писать 2,45%, когда там 2,45±1,50%, то есть как минимум между 0,95% и 3,95%. Потому и генетические дистанции при таких расчетах обречены на ложность, при вычитании одних малых чисел с большими погрешностями из других, да еще возводя в квадрат.
Короче, ясно, что мало у русских восточноазиатских гаплогрупп по сравнению с тремя основными — Н, U и U5 (и далее - J и Т), и этого достаточно. Остальных западно-евразийских тоже мало, на уровне единиц и долей процента. Ясно, что это случайно примкнувшие, и не нужно там пытаться «наводить науку» на сотых долях процента. Никакого научного смысла в такой точности нет. Главное, что это не имеет никакого прямого отношения к истории этнических русских. Эти доли процента могли попасть в указанные области когда угодно, хоть в этом веке, хоть в прошлом, хоть в позапрошлом.
Кстати, интересно, что гаплогруппа U, которую в изобилии нашли авторы, полностью отсутствует у этнических русских по другим данным, например, в известных таблицах Бехара. Она по данным Бехара и др. сохранилась только у узбеков, да и то на уровне менее 5%, а больше ни у кого в мире ее нет, хотя гаплогруппы U5 по тем же данным 10,6%. Так что здесь сильное противоречие с опубликованными ранее данными. Но не будем останавливаться, пусть авторы сами разбираются.
Дальше пошла любимая игра попгенетиков — все гаплотипы перемешали, невзирая на гаплогруппы, и построили поле «генетических дистанций», по прописи Кавалли-Сфорца 1967 года, 45 лет назад. То есть гаплогруппы-гаплотипы определяют по высшему классу, а затем впрягают в старую телегу. А смысл? Получили, что русские по всей солянке-совокупности всего и вся попадают в ближайшее окружение поляков, боснийцев, мордвы, марийцев, чувашей, татар и осетин. Можно добавить карелов и словенцев, они тоже недалеко. Ну, и что нового? Мы воздели в изумлении руки? Остается добавить, что это представители славянской, финно-угорской, тюркской и иранской языковых групп. Информативно, да?
Зато германская и балтская группы остались вдали, скажет торжествующе попгенетик. Вот именно это и показывает, что никакой научной ценности в этих построениях нет. Потому что половина литовцев — гаплогруппа R1a, общая с восточными славянами, как и пятая часть немцев, столько же у шведов, четверть у венгров, что тоже находятся в отдалении на диаграмме. А это и есть общая история, один род. Можно сказать, конечно, что у женщин одна корреляция, у мужчин — другая. Ну, и что это тоже даст? Что история женщин и мужчин у русских (и у других) разная? Да, наверное, и это тоже. Женщины в род приходили, или их силой привозили из разных краев, в том числе и из Восточной Азии. Ну и что это говорит об истории этнических русских?
«Русские ближе к волго-уральскому региону», делают вывод авторы из этой диаграммы. Что, рядом стоящие поляки, боснийцы и осетины — это волго-уральский регион? Что это, как ни откровенная натяжка? Впрочем, она объясняется легко. Следующая фраза: «Заметим, что это в полном согласии с их географическим расположением». Вот и подогнали. А то, что у поляков и русских по половине, а местами и по две трети одинаковой гаплогруппы R1a — это не близость географического расположения? То же и с белорусами. С каких пор белорусы — это волго-уральский регион?
Авторы здесь же делают ссылку на то, что «это позволяет узнать генетические характеристики сообществ, которые существовали до современных этнических групп», причем это ссылка на работу 1969 года одного из авторов, С. Рычкова. Это положение тоже непонятно в контексте данной работы. Что за «генетические характеристики» авторы узнали, которые существовали до современных этнических групп? Начнем с того, что мтДНК в этом контексте — это не «генетическая характеристика». Да и вообще, какой смысл здесь этой фразы? О каких «сообществах» здесь речь? О гаплогруппе H? Так она и сейчас существует.
Авторы представляют таблицу, в которой указаны «генетические расстояния» от усредненных русских мтДНК до усредненных мтДНК шести языковых групп. Вообще сама концепция приравнивать современные «языковые группы» к гаплогруппам-гаплотипам является более чем сомнительной. Иногда это работает, но для языковых и гаплогруппных изолятов (например, баски). Для балтов, например, у которых половина гаплогруппы R1a, это непонятно что дает. У авторов получилось, что ближе всех к русским — славянская языковая группа. Кто бы удивлялся, тем более, что у них преобладает опять же R1a (у мужчин) и Н (у женщин). Рядом — балты (причина та же). А дальше всех — иранцы, то есть осетины и персы. Опять, кто бы удивлялся. Тем более что это должно зависеть от времени, и от того, сравнивают с мужчинами или женщинами. Персы 2500 лет назад — это определенно преобладание R1a (у мужчин), ариев того времени. Это — родные братья наших праславянских предков той же гаплогруппы R1a. Кстати, и гаплотипы почти одни и те же, вплоть до 67-маркерных. Кто были их женщины — мне неясно.
Начиная с VII в. н.э. ариев значительно вытеснили мусульмане, гаплогруппы J2 в большой степени. Языки, тем не менее, остались иранские, хотя уже трансформировались в новейшие иранские языки. А современные осетины — это в большой степени гаплогруппа G (у мужчин). У женщин — надо разбираться, но судя по данным таблицы 3 — удаленные по мтДНК от русских женщин, что не удивительно. Ну и что эта таблица дает? Что осетинские женщины не сделали большого вклада в ДНК русских женщин? Я лично не удивляюсь. Думаю, что и никто не удивится. И что там «полностью подтверждает концепцию русского этногенеза», как утверждают авторы статьи? Неудивительно, что авторы не вынесли это в абстракт, там нечего выносить. Иначе пришлось бы написать, что русские женщины близки к балтийским и далеки от осетинок. Это, видимо, и есть «концепция русского этногенеза».
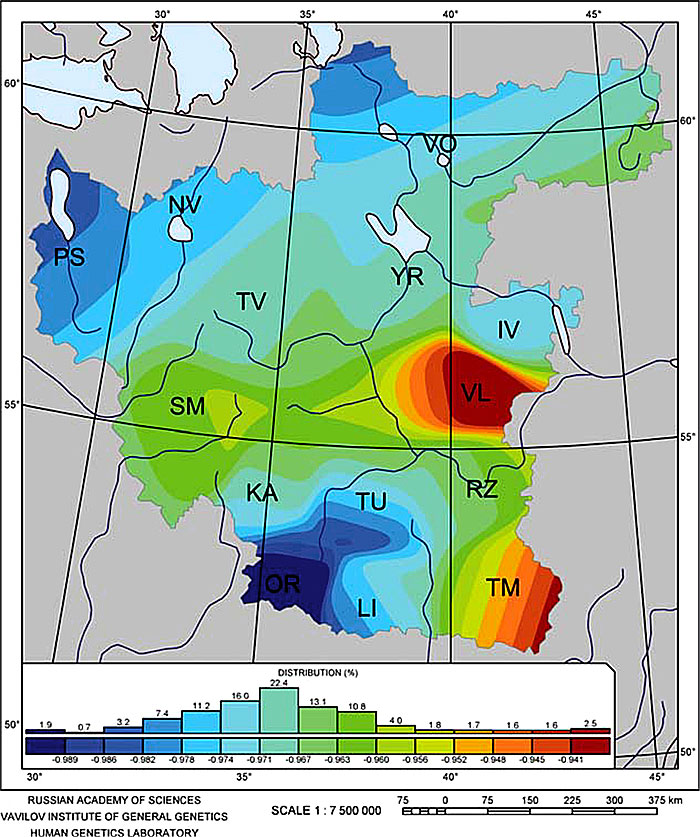
Авторы усреднили всех русских по «генетическим дистанциям» (про погрешности — см. выше), а затем по областям определили, какие области как отклоняются от «средних русских». Те, кто отклоняются меньше, изобразили светлым тоном, кто отклоняется больше — темным тоном. Авторы заметили, что то же самое разделение наблюдается и по Y-xpoмосомным «маркерам» (Гаплогруппам? Гаплотипам?). Для меня это большая новость, и я сильно сомневаюсь, что авторы понимают, что написали. Что там по гаплотипам-гаплогруппам отличается в Орловской и Владимирской областях — это какая-то мистика. Например, по данным, которые мы рассматривали в главах 7—9, в Орловской области из 42 гаплотипов 19 R1a, семь I, пять N1c1, четыре R1, четыре G, и по одному DE, J2, и К. Что там во Владимирской области может быть принципиально другое? Даже в среднем Орловская область такая же, как Россия, с 45% R1a (в России в среднем 48%) и 12% N1c1 (в России 14%).
Что в статье дальше, в отношении языка — это просто издевательство над здравым смыслом. Принцип такой, что любые цифры можно усреднять, складывать, вычитать, и почему это не сделать? Авторы усреднили «генетические дистанции» по северным, центральным, юго-западным и юго-восточным областям, и стали измерять усредненные же дистанции от мтДНК указанных выше шести языковых групп. Понятно, что дело здесь вовсе не в языках, а в географии. Получилось, ясное дело, что меньше всего расстояние от славян (потому что они и есть в основном славяне), равное -0,89±0,07 (это уже мои усреднения по их данным, но без включения погрешностей самих данных), далее — от балтов (литовцев и латышей), то есть от западно-прибалтийского региона -0,66±0,12, далее — от финно-угров (финны, эстонцы, карелы, венгры, мордва, марийцы, коми и удмурты) -0,62±0,36, далее — германская группа -0,27±0,40, тюркская (башкиры, чуваши, азербайджанцы, турки, узбеки, туркмены) +1,10±0,27, и иранская (осетины и персы) +1,33±0,18. Что это дает, кроме совершенно очевидного? Что это говорит об «истории этнических русских» — совершенно непонятно.
Чем дальше культурно и гаплогруппно, тем дальше и получается. А что, кто-то ожидал иначе? ЧТО с помощью полученных данных удалось показать? Какую историческую загадку решить? Какую новую методологию предложить и обосновать? И, главное, по названию статьи — что этим сказало об «этнической истории русских»? Что русские — не персы и не осетины? И не туркмены с азербайджанцами? Можно было бы отложить также религию в какой-либо цифровой форме, формы жилищ, похоронные ритуалы, облаченные в номера, форму букв алфавита, и получилось бы тоже самое. Наверное, все это говорит об истории в том смысле, что история была разной, раз алфавит другой — там арабская вязь, а там кириллица, а там латинские буквы, только разве в том была задача статьи?
На этом раздел «Результаты» закончился. Минуточку, а как насчет второй половины 1 тыс. н.э., к которому, по подаче абстракта, сводится «разнообразие» мтДНК? Об этом ведь пока не было ни слова, как и о «славянских племенах» и о колонизации ими Восточно-Европейской равнины. Приехали. Добро пожаловать в популяционную генетику. Как и предсказывалось, ничего об этом найдено не было, но чтобы было типа научно и неконфликтно, просто переписали из учебника для средней школы и подали, якобы это — ура! — подтвердили, причем строгими научными методами. Изучением мтДНК.
После этого раздел «Обсуждение» можно и не читать, все уже сказано. Там сейчас, разумеется, пойдет акробатика. Но мы прочитаем. Там особенно интересно.
Первое — что данные якобы показывают деление «исторических русских» (это-то откуда? Это ведь мтДНК современников при полном отсутствии расчетов датировок по мутациям в гаплотипах) на три части. Итак, это «северные русские», финно-угры, и «южные русские». Да, мы знаем, что про продвижении с севера на юг в Y-хромосомах в целом уменьшается доля N1c1 и возрастает доля R1a и I1/I2. Но N1c1 — это не «финно-угры», у тех совершенно другие гаплотипы, другие субклады этой гаплогруппы. Да и что там делить — «генетические дистанции» у «северных русских» (округленно, чтобы снять «наукообразность») 5,9; 3,8; 5,8; 5,8; у «юго-западных» 6,3 и 4,8, плюс 1,7 в Орловской области. Да нет в Орловской области такого различия от остальных юго-западных областей, там все перемешано. Такие различия надо по несколько раз проверять-перепроверять, а не просто бухнуть цифры, типа — смотрите, что у нас получилось. Дывитесь, громадные.
А вся разница всего лишь в том, что в Орловской области все три основные «русские» гаплогруппы — Н, U и U5 оказались по доле примерно такие же, как усредненные по России. Но поскольку авторы не рассчитывают погрешности, это сыграло с ними злую шутку. Давайте посмотрим. Погрешности пришлось рассчитать уже мне, авторы обходятся без них. Точнее, они приводят нечто (например, в таблице 3 в их статье), но там погрешности якобы на уровне сотых и тысячных долей процента. Это уже даже не смешно. Это — трагедия в отношении той же научной школы.
Ниже я рассчитывал погрешности при одной сигма (68% достоверности), но при двух сигма (95% достоверности) погрешности надо увеличить вдвое.
В Орловской области эту гаплогруппу имеют 51 человек из 115, то есть 44.3±6,2. Усредненные по России — 40,5±5,4.
В Орловской области эту гаплогруппу имеют 22 человека из 115, то есть 19.1±4.1. Усредненные по России — 20,8±6.0,
В Орловской области эту гаплогруппу имеют 12 человек из 115, то есть 10,4±3,0, Усредненные по России — 10,9±4,8.
Посмотрим на Калужскую область, она якобы резко отличается от Орловской (отличие Орловской области от средней по России по данным авторов 1,662, Калужской — 6,296).
В Калужской области эту гаплогруппу имеют 26 человек из 70, то есть 37,4±7,3. Усредненные по России — 40,5±5,4.
В Калужской области эту гаплогруппу имеют 18 человек из 70, то есть 25,7±6,1. Усредненные по России — 20,8±6,0,
В Калужской области эту гаплогруппу имеют 8 человек из 70, то есть 11,4±4,0. Усредненные по России — 10,9±4,8.
Как видно, Орловская и Калужская области одинаковы в пределах погрешности со средним по России по всем трем гаплогруппам — Н, U и U5. Но поскольку авторы погрешности не считали, а числа малые, то не знают, что погрешности были 14%, 21% и 29% для Орловской области, и 20%, 24% и 35% для Калужской. И это — для 68%-го доверительного интервала. Для 95%-го погрешности достигают 70%.
Вот и получается, что для юго-западного региона то, что показано как «девиации» 1,662 для Орла и 6,296 для Калуги — это на самом деле одно и то же в пределах погрешности экспериментальных данных. Поэтому все синтетические карты авторов и их выводы в отношении разных групп населения — рассыпаются как карточный домик, извините за каламбур.
Это же касается и все остальных цифр в статье. На самом деле распределение («разнообразие») мтДНК по всем 14 областям России одинаковое в пределах ошибки измерения. К этому же выводу я пришел еще в 2009 году в отношении распределения гаплогрупп Y-хромосомы по областям. Повторю — все, что можно сказать — это то, что к северу (от Пскова) доля гаплогруппы N1c1 возрастает, a R1a снижается, но по областям никакой воспроизводимой закономерности нет.
Все остальное обсуждение в статье уже не имеет никакого значения, так как строится на сплошной широкой ошибке. Все обсуждение построено на ложных цифрах и закономерностях, либо просто списано у археологов. Данные по самим мтДНК по областям полезны и могут пригодиться в будущем. Все остальное — нет.
Вот и вся популяционная генетика авторов.
Послесловие. Я направил копию этой главы основному автору обсуждаемой статьи, И.Ю. Морозовой в Институт общей генетики, и предложил высказать свои соображения «в порядке дискуссии». И.Ю. Морозова вежливо отказалась.
Статья опубликована относительно недавно, в журнале «Генетика» (Тетушкин, 2011). Представляет вполне добротное описание истории этой области науки, начиная от Дарвина, через Н. Кольцова и Ю. Филипченко и до недавнего времени. ДНК-генеалогия в статье, естественно, не описана, хотя несколько ссылок под запятую даны, в глухом, так сказать, варианте. Причина опять понятна — автор не владеет методологией и материалом ДНК-генеалогии, поскольку она не «генетическая генеалогия» и не популяционная генетика, и автор обсуждаемой статьи это фактически подтвердил.
То, что автор не владеет материалом ДНК-генеалогии, он продемонстрировал тем, что по ходу описания задел три вопроса, которые популяционные генетики как не понимали раньше, так не понимают и сейчас. Это (1) история с «модальным гаплотипом коэнов», (2) история с «популяционными скоростями Животовского», и (3) история с «мутациями в парах отец-сын». Разберем эти три случая в показательном, так сказать, варианте.
По мнению автора рассматриваемой статьи, определяющее значение для формирования идеологии и методологии «генетической генеалогии» в ранний период ее развития сыграли, в частности, публикации о Y-хромосомах еврейских священников, коэнов. С этим можно согласиться, но в двойственном варианте. С одной стороны, столь броская тема, поданная в стиле почти «желтой прессы», несомненно, привлекла внимание общественности к «генетической генеалогии». С другой стороны, то, как это обрабатывалось в научном отношении, заложило основы фантазийного и научно несостоятельного, некритичного подхода «генетической генеалогии» до настоящего времени, что, собственно, и является предметом нашего анализа. Это тут же породило и якобы 30-тысячелетнюю историю гаплогруппы R1b в Европе, и «украинское убежище гаплогруппы R1a во время ледникового периода», и «прародину» гаплогруппы R1a в южнорусских степях, назвав ее при этом почему-то «украинской» гаплогруппой.
Примерно то же произошло и с «модальным гаплотипом коэнов». То, что этому «модальному гаплотипу» как минимум 9 тыс. лет — авторы не знали и не подумали, в попгенетике альтернативных вариантов вообще не признают. Но это еще полбеды. А вот то, как анализировали данные, это уже хуже. Рассмотрим это.
В 1997 году в журнале Nature была опубликована короткая статья «Y-хромосомы еврейских священников» (Skorecki et al, 1997). Эта статья вскоре приобрела статус пионерской и легендарной. В ней было объявлено, что частота определенных гаплотипов еврейских священников отличается от частоты встречаемости гаплотипов «простых» израэлитов. В работе с помощью двух маркеров ДНК изучались гаплотипы 188 евреев из Израиля, Северной Америки и Британии. При этом те, кто не был уверен в своем коэнском происхождении, включая левитов, выводились в отдельный список «израэлитов». Так были отобраны 44 коэна-ашкенази, 24 коэна-сефарда, и 120 не-коэнов, израэлитов. Авторы, впрочем, не обмолвились, что доля коэнов в выборке (36%) намного превышает ожидаемую цифру 5%, приведенную в самом начале статьи.
В итоге авторы сообщили, что нашли ДНК-метку, которая встречается только у 1,5% коэнов, но у намного большего числа (18%) израэлитов. Другая метка, не из Y-хромосомы, взятая для сравнения, встречалась в одинаковой степени у коэнов и израэлитов. Коэны, стало быть, нормальные люди (так это читается у авторов), но отличаются меткой в Y-xpoмосоме от других евреев.
Курьез статьи в том, что ее главное положение, что коэны отличаются по метке в Y-хромосоме ДНК от остальных евреев, относится к 36% евреев в выборке, которые коэнами по своей доле быть никак не могут. Перефразируя авторов, данные статьи показали, что треть всех евреев, НАЗЫВАЮЩИХ себя коэнами, генетически (в смысле наследственно) отличаются от остальных евреев.
Поскольку в статье использовалась устаревшая номенклатура и гаплотипы не идентифицировались и с тех пор эти данные не перепроверялись, то сейчас трудно сказать, в чем же состояли отличия коэнов от израэлитов, которые подчеркивали авторы статьи, тем более различия более чем на порядок измеряемой величины. По современным данным таких выраженных отличий нет. Но дело было сделано, внимание к вопросу привлечено.
Через год в том же журнале вышла вторая статья на ту же тему, с заметно измененным авторским коллективом. Статья называлась «Происхождение ветхозаветных священников» (Thomas et al, 1998). В развитие высказанных ранее идей, авторы «логическим путем» обосновывают положение, что гаплотипы коэнов и левитов должны отличаться от гаплотипов других евреев и иметь общего предка не ближе, чем примерно две-три тысячи лет назад. Затем авторы рассматривают гаплотипы евреев и заключают, что с левитами эта логика не проходит, но гаплотипы коэнов действительно имеют общего предка примерно 3000 лет назад.
В статье рассматривались 6-маркерные гаплотипы (DYS 19, 388, 390, 391, 392, 393) и шесть снипов (устаревшая или уже забытая номенклатура) в Y-хромосомном материале 306 евреев из Израиля, Канады и Англии, и было найдено 112 вариантов гаплотипов. Только один — получивший наименование «модальный гаплотип коэнов» (МГК) — имел наибольшую частоту у коэнов, как среди коэнов-ашкенази (45%), так и среди коэнов-сефардов (56%). МГК плюс МГК с одной мутацией наблюдался у 69% и 61% коэнов ашкенази и сефардов, соответственно. У израэлитов эти частоты были 13% (15%) и 10% (14%), соответственно (в скобках — частоты МГК с одной мутацией в шести маркерах).
Авторы оценили время, когда жил первопредок коэнов, исходя из отклонений величины аллели от средней по каждому маркеру, принимая среднюю скорость мутаций 0,0021 на маркер на поколение. Расчет проводился по пяти маркерам, маркер 388 был отброшен по причине его отклонений от «ступенчатого характера мутаций» (непонятно, почему и в чем это выражалось, но в статье это не указано). Поскольку для всех евреев в выборке это среднее отклонение было равно 0,226 мутаций на маркер, то было рассчитано, что общий для всех 306 евреев предок жил 106 поколений, или 2650 лет назад, принимая 25 лет на поколение. Авторы замечают, что при 30 годах на поколение получится 3180 лет до общего предка. Для ашкеназов и сефардов отдельно эти величины были равны (25 лет на поколение) 2619 и 2684 лет, соответственно.
К этохму можно заметить, что за прошедшие со времени публикации десять лет величины скоростей мутаций были уточнены, и для рассматриваемых пяти маркеров скорость мутации равна 0,00171 на поколение. Тогда общий предок коэнов, по данным авторов, жил 0,226/0,00171=132 поколения назад без поправки на возвратные мутации, то есть 152 поколения с поправкой, или 3800 лет назад, а еще точнее 3800±430 лет назад. А если брать 30 лет на поколение, то общий предок коэнов жил 4560±520 лет назад, то есть во времена Авраама или даже раньше.
В общем, можно считать, что полученные величины не противоречат библейским данным. Но с этим подходом есть проблемы, и принципиальные. Менее серьезной является проблема, что само введение понятия «модальный гаплотип коэнов» было неудачным, поскольку он наблюдается не только у коэнов и не у всех коэнов, но и у многих израэлитов, а также у многих арабов и вообще у многих носителей других гаплогрупп, в первую очередь J1 и J2. Многие коэны, имеющие данный гаплотип, относятся к одной из этих гаплогрупп, что в свою очередь, формально говоря, практически исключает происхождение тех или других от одного предка, звали его Аарон или как-то иначе. Наконец, этот гаплотип со временем искажается мутациями, что автоматически выводит его из понятия «гаплотип коэнов». В общем, с «модальным гаплотипом коэнов» так много исключений, что не видно самого правила. На самом деле его и не существует.
Самое главное, что сама концепция расчета общего предка, используемая в статье, в принципе порочна. Как было нами показано еще пять лет назад, подавляющее большинство евреев, носителей «гаплотипа коэнов», являются потомками по меньшей мере двух (!) относительно недавних предков, один или более из которых определенно жили уже в нашей эре, во времена Диаспоры. То, что сделали авторы обсуждаемой статьи — это просто усреднили «отклонения величины аллелей от средней» для двух разных предков, а на самом деле от нескольких, каждый из которых имеют свои генеалогические линии, потому что в своих расчетах разных предков не разделяли.
Фактически авторы измеряли «среднюю температуру по больнице». При этом время жизни «общего предка» могло получиться любое. Получилось 2650 или 3180 лет, если брать 25 или 30 лет на поколение. А взяли бы другое количество лет на поколение — получились бы другие цифры. И проблема даже не в том, что усредняли по разным предкам, а в том, что потомки этих разных предков, разных ДНК-генеалогических линий, были представлены в выборке в разных количествах. Поэтому те, кто были наиболее многочисленны в выборке, «тянули» время рассчитываемого предка на себя. Именно потому могла получиться почти любая (в широких пределах) цифра времени жизни «общего предка», на самом деле фантомного.
Рассмотрим, как на самом деле надо считать. Список из 194 гаплотипов евреев гаплогруппы J был опубликован в работе Бехара и др. (Behar et al, 2003). 91 из них были идентичны друг другу:
Это и есть тот самый «Модальный гаплотип коэнов» (МГК). В общем, уже ясно, что когда базовый (предковый) гаплотип находится в серии в количестве 91 из 194, то есть половина, то предок явно не древний. Тем не менее, авторы цитированных выше работ в журнале Nature в 1997 и 1998 годах объявили, что МГК появился больше 3 тыс. лет тому назад. Этого просто не может быть, глядя на эти гаплотипы и обращая внимание на частоты встречаемости гаплотипов в серии.
Полный набор из 194 гаплотипов содержит 263 мутации от «модального гаплотипа коэнов», то есть указывает на общего предка, жившего 263/194/0,0088= 154->182 условных (по 25 калиброванных лет) поколения назад (стрелка — поправка на возвратные мутации), то есть 4550±530 лет назад. Здесь 0,0088 — средняя скорость мутаций для 6-маркерных гаплотипов. Но посмотрим на долю остаточных базовых гаплотипов: [ln(194/91)]/0,0088=86→95 поколений, 2375±340 лет назад. Разница до «одного и того же общего предка» — более двух тысяч лет. Так быть не может. Значит, там не один предок, а больше. Нельзя так считать, как считали авторы статей в Nature. И как считают, к сожалению, в десятках других статей, написанных попгенетиками. А то, что одного общего предка быть не может, показывает дерево гаплотипов. Видно, насколько дерево гетерогенно.
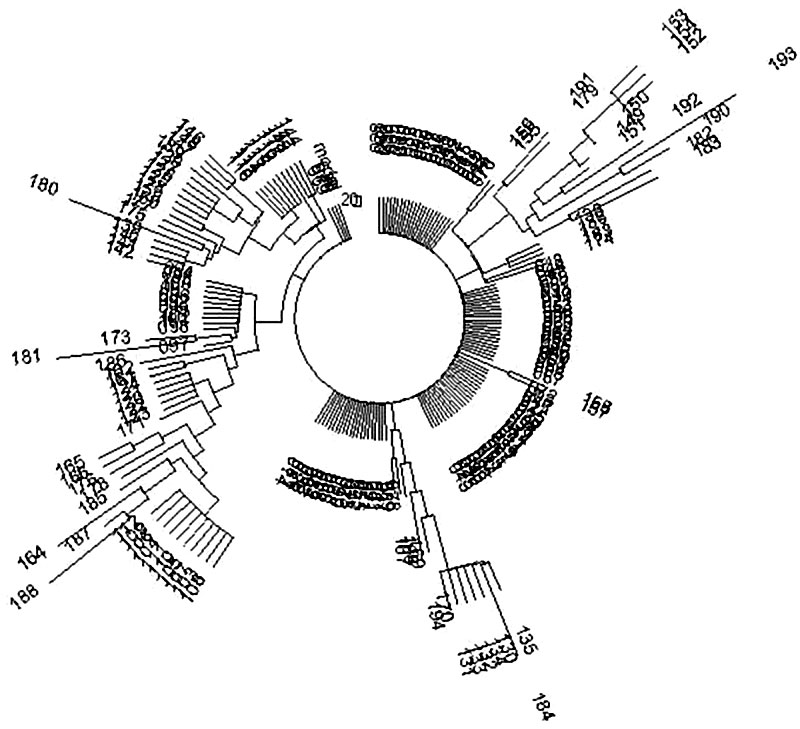
Посмотрим на другую выборку, гаплогруппы J1 евреев, извлеченную из базы данных YSearch. 90 гаплотипов, в них 41 базовый и 123 мутации от точно такого же базового гаплотипа. Это дает 123/90/0,0088=155 поколений до общего предка (а в «научной выборке» было 154 поколение!) и ln(90/41)/0,0088=89 поколений («научная» выборка дала 86 поколений), без поправок на возвратные мутации, которые уже не нужны, поскольку речь о «фантомном» предке. Но в любом случае, «коммерческая» и «научная» выборка дали одни и те же результаты. Обе показывают, что предок был не один. Но обе показывают, что распределение данных ДНК-генеалогических линий у евреев является стабильным и воспроизводимым. Вот только рассматривают их авторы публикаций неверно.
Детальное изучение выборок показало, что эти выборки содержат по меньшей мере восемь (!) генеалогических линий, каждая из которых берет начало от своего общего предка, и предки эти жили (без погрешностей расчетов) 11100, 6200, 5100, 3500, 1800, 775, 1650 и 1350 лет назад. Последние две линии — «Модальный гаплотип коэнов», которых оказалось два уже в 12-маркерных гаплотипах.
Далее оказалось, что картина с «модальным гаплотипом коэнов» становится еще более курьезной, поскольку такой же гаплотип обнаружился и в другой гаплогруппе, J2. Так, в работе Бехара и др. (Behar et al, 2004) была опубликована выборка из 88 гаплотипов евреев гаплогруппы J2, 25 гаплотипов из которой были идентичны друг другу:
По чистой случайности этот 6-маркерный гаплотип идентичен «модальному гаплотипу коэнов» гаплогруппы J1. Эта идентичность вызвала массу дискуссий, которые продолжаются и сейчас — почему идентичны гаплотипу коэнов, ведь это же другая гаплогруппа! А это просто причуды статистики. Случайно именно эти шесть аллелей совпали, а многие из остальных заметно различаются в «гаплотипах коэнов» гаплогрупп J1 и J2. Например, на первых 25 маркерах между ними 11 различий.
Все 88 гаплотипов содержат 160 мутаций от базового гаплотипа. По доле базового гаплотипа число поколений до общего предка равно ln(88/25)/0,0088=143 поколения, или 167 поколений с учетом возвратных мутаций, то есть 4175±930 лет назад, по мутациям — 160/88/0,0088=207 поколений, или 260 поколений с учетом возвратных мутаций, то есть 6500±830 лет. Опять разнобой, более двух тысяч лет разницы между якобы одним и тем же общим предком, значит — более одного общего предка. Но это и так видно, что в дереве гаплотипов — явно не менее трех ДНК-генеалогических линий («плоские» ветви), значит, считать их возраст надо по индивидуальным ветвям.
Проверим по другой базе данных, по другой выборке, «коммерческой». В ней — 75 гаплотипов евреев гаплогруппы J2, которые включают 15 базовых гаплотипов, таких же, как и в «научной» выборке, и содержат 135 мутаций. Это дает ln(75/15)/0,0088=183 поколения и 135/75/0,0088=205 поколений до общего предка (без поправки на возвратные мутации). Опять более одного общего предка, на что указывает и дерево гаплотипов.
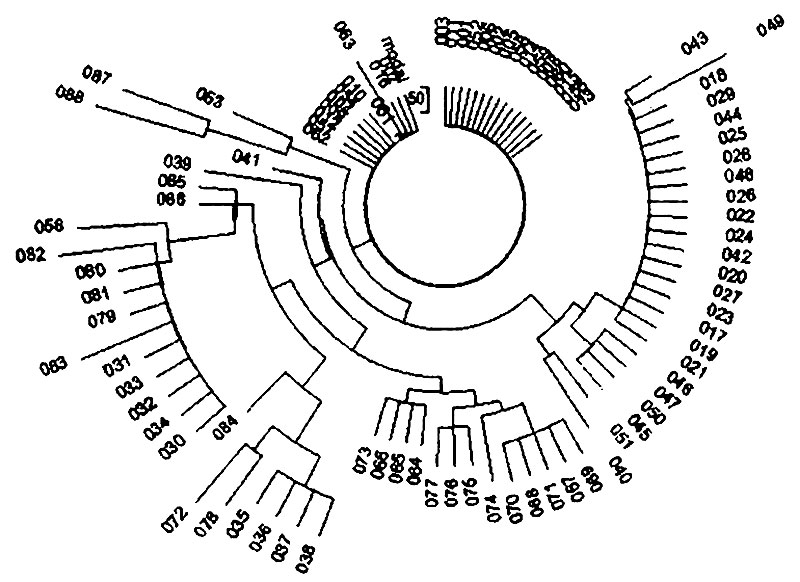
Анализ деревьев гаплотипов показал, что у евреев гаплогруппы J2 имеется по меньшей мере пять общих предков. Те, от которых произошло наибольшее количество современных евреев гаплогруппы J2, жили 6100, 900, и 1200 лет назад (без учета погрешностей). Последняя генеалогическая линия — «модальные гаплотипы коэнов».
Эти примеры опять показывают, что нельзя рассчитывать времена до общего предка, основываясь только на числе мутаций в гаплотипах, без приложения других критериев, которые показывают, «чистая выборка» или нет, то есть происходит ли она от одного общего предка или нет.
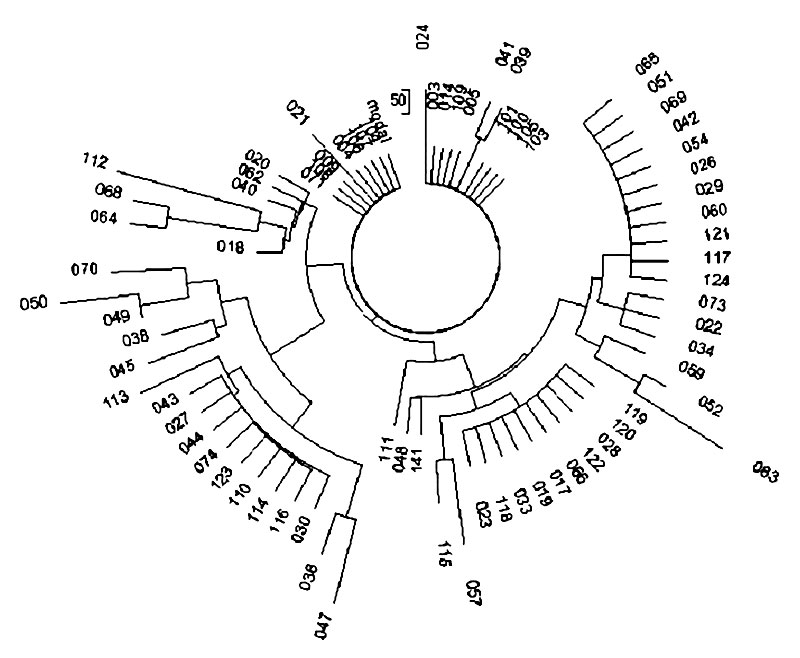
Вопрос с гаплотипами коэнов был проанализирован в наших работах, опубликованных в журнале Human Genetics (2009), и показано, что наиболее многочисленная группа потомков коэнов гаплогруппы J1 происходит от общего предка, который жил 1075±130 лет назад или 1050±190 лет назад, по двум разным выборкам, первая «академическая», вторая «коммерческая», по 67-маркерным гаплотипам.
Возвращаемся к рассматриваемой статье Е.Я. Тетушкина. Он пишет: Scorecki et al... показали, что основная часть священнослужителей (коэны) имеют идентичные Y-STR-маркеры, которые могли быть унаследованы от первосвященника Аарона (брата Моисея), который, согласно Библии, жил более 3000 лет назад (позднее, оценки частот этих маркеров у современных коэнов были значительно подправлены в сторону их уменьшения) [обратный перевод].
Это называется — начал за здравие, кончил за упокой. Так могли быть унаследованы или «значительно подправлены», что ломает все, что сказано выше? Мало того, что фраза совершенно неинформативна, поскольку не сообщает, что значит «подправлены», на каком основании подправлены, к чему это приводит. Она, что называется, «и вашим, и нашим». Хорошая наука, неконфликтная — только и остается повторить. Вроде и так, и вроде не так, а что именно — то ли не знаем, то ли умолчим.
В обсуждаемой работе автор, Е.Я. Тетушкин, немалое внимание уделил «проблеме» со скоростями мутаций, «популяционной», или «эволюционной», и «генеалогической», которые «значительно различаются». На самом деле никакой проблемы там нет, если понимать, что первая «скорость» имеет тот же смысл, что «свалить все в кучу, усреднить и поделить на что получится», то есть никакого смысла на самом деле не имеет, а вторая применяется попгенетиками настолько неразборчиво, что имеет смысл только при случайных сериях гаплотипов, не имеющих ветвей, или отдельных ДНК-генеалогических линий, каждая со своим отдельным общим предком.
В качестве аналогии можно привести пример из химии, когда в растворе есть смесь химических реагентов, которые беспорядочно реагируют друг с другом. Пытаться описать это некой единой «константой скорости реакции» (типа «эволюционной» или «популяционной») — это не понимать сути химии. В химии само понятие «константа» в подобных случаях имеет смысл, что по мере исчерпания реагента скорость остается пропорциональной концентрации вещества (v=kc), и константа к — это и есть показатель пропорциональности. В более общем случае для константы скорости первого порядка это записывается как c/c0 = е-kt, или ln(c0/c)= kt, и здесь опять k — это константа скорости реакции, показатель пропорциональности левой и правой части приведенного уравнения. Это же уравнение обязано описывать и накопление мутаций в гаплотипах, и уменьшение количества базовых, или предковых гаплотипов в серии гаплотипов, и константа скорости мутаций обязана быть одной и той же, на то она и константа. Именно потому основным критерием в ДНК-генеалогии является совпадение величины константы скорости мутации, определяемой как по накоплению мутаций («линейный метод» расчета), так и по уменьшению количества базовых гаплотипов в серии («логарифмический метод» расчета, при котором мутации вообще не считаются).
В «популяционных» расчетах по скоростям мутаций такого нет и близко. Там берется общее количество суммарно набежавших мутаций, от разных ветвей разного возраста и происхождения и от разных предков, вся эта мешанина делится на количество гаплотипов, что и называется некой «вариацией», «разнообразием», и далее делится на некую «популяционную» скорость мутаций, вычисленную из некой абстрактной системы, не имеющей никакого отношения к реальности. Проверить-то все равно нельзя, никаких критериев проверки нет, что получится, то и получится. А разделить что-то на что-то всегда можно. А именно, в одном из вариантов («метод Животовского») берется 10 тысяч гаплогрупп (!) за все время существования человечества (!), то есть принимается идеальное перемешивание системы гаплотипов по всему человечеству во времени и пространстве. Тогда расчеты показывают, что для такой системы идеально усредненная константа скорости мутации при определенных приближениях равна 0,00069 мутаций на локус за 25 лет. При этом все локусы считаются одинаковыми, и возвратных мутаций нет, как нет и их учета. Такого на самом деле никогда не бывает.
Все это приводит к тому, что подобные расчеты применять на практике просто нельзя. А попгенетики их бездумно применяют сплошь и рядом для любых систем — и те, которые случайно оказались состоящими из одной ветви, и те, которые состоят из двух-трех ветвей, которые можно (и нужно) было бы разделить, и вообще для любых систем гаплотипов. В итоге результат оказывается совершенно непредсказуемым и потому бессмысленным.
Автор обсуждаемой статьи, Е.Я. Тетушкин, сути этого не понимает, хотя, похоже, ощущает, что что-то здесь не то. Он пишет, что согласно Животовскому и др. (Zhivotovsky et al, 2004), «средняя популяционная скорость мутаций» для Y-хромосомных STR принимается за 0,00069 мутаций на маркер на поколение, и что «генеалогическая» скорость мутаций, по мнению многих авторов, «намного выше», и что «эта сложная ситуация требует специального рассмотрения». Забавно то, что Е.Я. Тетушкин читал мою статью, в которой это «специальное рассмотрение» было специально проведено, и на нее ссылается, но то ли ее не понял, то ли понять не захотел. Это следует из того, что он пишет, что работа Животовского по «популяционным скоростям» критиковалась непрофессионально.
Давайте разберемся, у кого там непрофессионально. В ответ на мою критику в журнале Human Genetics (Klyosov, 2009а) работы (Hammer, 2009а) Хаммер с Животовским пишут в ответ (Hammer, 2009b): «By examining Y-STR variation within Y chromosome haplogroups (i.e., defined by unique event polymorphisms) in populations with documented short-term histories, they inferred an evolutionary effective mutation rate of 0,00069 per 25 years on average... (Zhivotovsky et al, 2004).... (i.e., the effective rate was calibrated for time periods of ~ 1,000 years). Since 2004, this approach has been employed by the researchers and critically evaluated in the scientific literature (e.g., Arredi et al. 2004; Di Giacomo et al. 2004: Gayden et al. 2007; Sengupta et al. 2006; Xue et al. 2005; Zhivotovsky and Underhill 2005)». Перевод: «При изучении вариаций в гаплотипах Y-xpoмосомных гаплогрупп (то есть обусловленных снипами) в популяциях с документированными непродолжительными хронологиями, они пришли к выводу об эволюционных эффективных скоростях мутации 0,00069 за 25 лет... (Zhivotovsky et al, 2004). ...(то есть эти эффективные скорости были калиброваны за период времени примерно 1000 лет). С 2004 года этот подход был применен исследователями и критически рассмотрен в научной литературе (например, Arredi et al. 2004; Di Giacomo et al. 2004: Gayden et al. 2007; Sengupta et al 2006; Xue et al. 2005; Zhivotovsky and Underhill 2005).
На самом деле, как будет показано ниже, никакой «калибровки» в исходной работе Л. Животовского (Zhivotovsky et al, 2004) не было. Были приведены нестыкующиеся данные, или данные вообще не приведены, и затем серией произвольных манипуляций часть данных убиралась, пока «скорости мутаций» не достигли одного порядка (именно так), которые были обозначены на «0,00069 мутаций на маркер на поколение» с погрешностью того же порядка. В последующие пять лет эта работа постоянно цитировалась с указаниями, что данные «калибровались» и «критически проверялись», пока в академическом мире в это сами не уверовали. Никакой «критической проверки», поддерживающей «коэффициент Животовского» в литературе тоже не было. К сожалению, метод продолжает применяться, давая совершенно неверные и непредсказуемые данные и приводя к совершенно ложным интерпретациям. В наших работах те же серии гаплотипов были перепроверены, и получены совершенно другие скорости мутаций, согласующиеся с нормальной «генеалогической» скоростью мутаций.
Можно между делом отметить, что из цитированных Хаммером, Животовским и др. шести работ, которые «критически рассматривали», в трех соавтором был сам Л. Животовский (Gayden et al. 2007; Sengupta et al. 2006; Zhivotovsky and Underhill 2005), а остальные метод и не обсуждали, а просто бездумно использовали, так что какое уж там «критическое рассмотрение». Но здесь ситуация более «подтасовочная», чем просто бездумное использование, потому что никакой «калибровки» Л. Животовским с соавторами своей скорости 0,00069 не было и близко. Были неопределенные манипуляции с удалением некоторых гаплотипов из рассматриваемой серии, были некоторые неопределенные же пассы, которые в итоге привели к цифрам с огромными погрешностями, под которые подпадает практически любой диапазон скоростей мутаций. Говорить о «калибровке» именно величины 0,00069 — это, по меньшей мере, недобросовестность. Ниже я покажу в деталях, что там была за «калибровка».
Это — не просто демонстрация примитивного, неверного и фактически необоснованного подхода, который Л. Животовский активно насаживает в совместных публикациях по всему миру. Хуже, когда этот подход подается как «калиброванный», причем подается самим Л. Животовским, который прекрасно знает, что никакой калибровки там не было. Еще хуже, что этот метод тиражируется в «академической» литературе. В статье (Athey, 2009), Whit Athey пишет (перевод): «Популяции с древними корнями подвержены эффекту популяционной динамики, то есть исчезновение многих линий и заметная экспансия других линий, плюс возможное уменьшение продолжительности поколений (почему в такой же степени не уменьшение? — А.К.). Все эти эффекты требуют корректировочных факторов, которые совместно могут достичь фактора, равного двум, который использует Животовский в своей часто цитируемой статье (Zhivotovsky, 2004).
На самом деле Л. Животовский использует фактор, примерно равный трем, но Athey сам начинает пускаться в манипуляции, переводя «фактор Животовского» из 0,00069/25 лет в фактор 0,00092/33,3 года, и беря одну из многих величин для скорости мутации для пар отец-сын 0,00184 (ссылку на которую он даже не дает), и получая фактор два.
Иначе говоря, идет активное использование и цитирование подхода, за которым ничего не кроется, кроме недоразумений и подтасовок.
В чем, собственно, метод состоит? Рассмотрим подробнее.
Если бы не было «генетического дрейфа», вызываемого природными катаклизмами, войнами, мором и другими причинами массовых вымираний, а также другими вариантами вымирания племен, родов и прочих популяций, а также переездом носителей гаплотипов на новые территории и начала (точнее, продолжения, но часто со сдвигом) там новых линий, то дерево гаплотипов было бы вполне симметричным и однородным, как, например, показано ниже для субкладов Rlb-P312 и Rlb-L21. Все несколько сотен гаплотипов каждого из этих и подобных деревьев сходятся к одному общему предку, который в показанных сериях жил 3950±400 и 3600±370 лет назад.
Но такие случаи редки, да и время здесь относительно небольшое, менее 4000 лет назад. Тем не менее, «метод Животовского» не дает рекомендаций, на каких временных показателях его применять. Хаммер с Животовским в цитированной выше статье («Response») считают, что этот метод следует применять при временах примерно 1000 лет назад, и, естественно, ранее. В данных двух случаях «метод Животовского» дал бы для популяций субкладов R-P312 и R-L21 времена общего предка 9030 и 8300 лет назад. При таких величинах интерпретация исторических событий была бы совершенно сдвинута. Вот так она и оказалась сдвинутой в недавней работе Андерхилла и Животовского с сотр. (Underhill et al, 2009), в которой времена жизни предков популяций субклада R1a1a7* 2600 лет назад были представлены как 8000 лет назад (как раз обычное 300%-ое завышение этих авторов), общие предки русских гаплогруппы R1a1a*, как «оказалось», жили 8700 лет назад, иранцев — 9400 лет назад, поляков — 11300 лет назад, кавказцев — 12200 лет назад, и так далее. Мало того, что эти цифры не имеют никакого отношения к реальности, но на их основании авторы опять — в который раз! — утверждают, что «никакого «потока» гаплогруппы R1a из Восточной Европы в Азию, в том числе в Индию, не было, во всяком случае, со времен среднего голоцена» (то есть за последние 7—8 тыс. лет).
Остается поражаться не только сплошным ошибкам авторов (Underhill et al, 2009), но и отсутствию их представлений об археологии, о том, что уже давно опубликованы данные о нахождении ископаемых R1a в андроновской культуре (Северный Казахстан, Южный Урал, Южная Сибирь). О том, что гаплотипы R1a у этнических русских и индийцев практически идентичны. Иначе говоря, грубые ошибки Л. Животовского и его соавторов в методологии расчетов переходят уже в системное искажение истории.
Еще пример. Приведенные выше величины 3950±400 и 3600±370 лет назад показывают, что в те времена имело место равномерное и неуклонное заселение Европы носителями гаплогруппы R1b1b2. Интересно, что в те же самые времена популяция R1a практически исчезает из Европы, и вновь заселяет Европу только 2600—3000 лет назад и позднее, а именно 2725±300 лет назад для центрально-европейской ветви R1a, 2575±300 для западнославянской ветви, 2800±350 для северо-карпатской ветви, 2750±370 для западно-евразийской ветви, 2600±290 для балто-карпатской ветви, 2550±320 для южной евразийской ветви, 2150±300 для западно-карпатской ветви, и так далее. «По Животовскому», первые R1a в Европе должны были появиться не 10—12 тыс. лет назад, а 25—30 тыс. лет назад, что противоречит здравому и историческому смыслу, в той же степени, в какой гаплогруппа I1 должна была появиться в Европе не 21400 лет назад, а 60 тыс. лет назад. И это только I1, не говоря об I2 и родительской I.


Возвращаемся к равномерным, симметричным деревьям. Ясно, что «метод Животовского» по своей сути к ним не подходит. Никаких генетических дрейфов там не выражено, четко выявляется один общий предок, и никаких оснований занижать скачком скорость мутации нет.
В древних популяциях, как правило, из-за вымирания многих ДНК-генеалогических линий, дерево гаплотипов выглядит как набор «обрубков» ветвей. Это обрубки — результат сдвигов в базовых гаплотипах, результат «бутылочных горлышек» популяции. У каждой из этих ветвей есть свой общий предок со своим базовым гаплотипом. Из набора этих базовых гаплотипов рассчитывается время жизни общего предка для всего дерева.
Например, дерево гаплотипов гаплогруппы Т Армении состоит из нескольких ветвей, у каждой свой базовый гаплотип, и две самые большие плоские ветви (слева внизу и справа наверху) имеют времена жизни общих предков примерно 500 и 1000 лет назад. Общий предок обеих ветвей жил примерно 7400 лет назад. Скорости мутаций при этих расчетах были ранее откалиброваны по известным генеалогиям и равны 0,034 мутаций на гаплотип на условное поколение (25 лет), или 0,002 мутации на маркер на поколение. Расчет «чохом» по всему дереву показал наличие 246 мутаций от суммарного базового гаплотипа дерева, что дало 246/35/0,034=206→258 поколений, то есть 6450±770 лет до общего, «фантомного» предка всего дерева. Формально это в пределах погрешности расчетов с величиной, рассчитанной по двум противоположным ветвям дерева. Более детальный анализ того же дерева по всем четырем ветвям (Лабай, 2011) через месяц после публикации (Клёсов, 2011) показал, что общий предок всего дерева жил примерно 6760 лет назад, то есть в согласии с предыдущей работой. В данном случае совпадение расчета «чохом» и по отдельным ветвям было вызвано довольно хорошей симметричностью дерева, в котором «веса» ветвей уравновешивали друг друга. Но если приложить «метод Животовского», то получится 246/35/17/0,00069=599 поколений по 25 лет, то есть 14975 лет до общего предка, завышение на 220%. Естественно, была бы приведена огромная погрешность, где-то от 5 тыс. до 40 тыс. лет назад, и вопрос бы считался решенным.
При этом мало того, что получена ложная информация, но и потеряна ценная информация об истории отдельных ветвей дерева, об их базовых гаплотипах, два из которых принадлежали недавно живущим общим предкам молодых популяций Армении. То есть полностью искажается и теряется информация об истории населения страны. Поразительно, что популяционные генетики это не только не понимают, но и не хотят понимать.
Читатель может подумать, что данные «по Животовскому» всегда получаются завышенными. Ничего подобного. Они могут оказаться любыми. Никакой связи с реальностью они не имеют. В зависимости от числа гаплотипов и формы дерева они могут гулять по любому мыслимому диапазону. Вообще все расчеты Л. Животовского и тех, кто имеет несчастье использовать его «метод», можно свести к одной фразе — «это происходило давно». Во всяком случае, это будет точнее и научнее.
Далее, при таком «счете по Животовскому» игнорируется, что гаплотипы разной длины имеют разную среднюю скорость мутаций, что нужно вводить поправку на возвратные мутации, которая прогрессивно возрастает с увеличением времени до общего предка, что предок может быть один, или ветвей может быть несколько, что среди них могут быть совсем недавние ветви, и могут быть ветви дочерние от других ветвей самого дерева. Все это игнорируется, и делится на некий постоянный для всех случаев фактор. Он просто призван «удревнить предка», насколько — не важно. Все равно никто не проверит.
Проверили. Оказалось, метод Животовского — одна большая сплошная ошибка. Он ведет «счет» как древние индейские племена: один, два, три, а что выше — «много». Вот у него и есть «много», а сколько — неважно.
Да, но как же «калибровка»? Взглянем, какая там была калибровка.
Открываем «часто цитируемую статью» (Zhivotovsky et al, 2004). Читаем для начала резюме статьи. В нем — две фразы. В первой фразе резюме сообщает, что «Мы установили эффективную скорость мутации на усредненном тандемном локусе Y-хромосомы 0,00069 в расчете на 25 лет, со стандартным отклонением 0,00057». Для начала неплохо. Правда, в академических статьях цитируя «0,00069», не цитируют, что «стандартное отклонение» равно почти 100%, точнее, 83%. То есть в диапазоне от 0,00126 до 0,00012, более чем в десять раз. Величина 0,00126 уже близка к 0,00147 для 6-маркерных гаплотипов, с которыми Животовский в той статье и работал, только 14% разницы. Наверное, эту цифру и получил бы, если бы не манипулировал с гаплотипами по своему произволу. К этому мы еще вернемся.
Вторая фраза в резюме: «Это значение мы использовали для определения времен экспансии африканских банту, дивергенции полинезийских популяций (маори, островов Кука, и самоанцев), и происхождения популяции цыган из Болгарии».
Посмотрим, как и что они использовали.
Раздел «Материалы и методы». Смотрим про африканских банту. Читаем — «анализировали 148 человек гаплогруппы Е3а7-М191», перечисляются племена, сообщается, что анализ проводили по 10-маркерным гаплотипам.
Раздел «Результаты». Данных по банту нет, они вообще в данном разделе не упоминаются.
Раздел «Обсуждение». Банту не упоминаются.
Раздел «Применение». Говорится, что применили полученную величину 0,00069 к популяции банту, чтобы посмотреть, как это коррелирует с археологическими, лингвистическими и историческими данными. Далее говорится, что если усреднить по всем 11 племенам и популяциям, перечисленным в разделе «Материалы и методы», то получится 3400±1100 лет до «экспансии» банту в подгруппе со снипом М191. И далее: «Археологические и лингвистические данные поддерживают это значение, поскольку показывают присутствие банту в Западной Африке в неолитические времена, ~1000 лет до н.э., или даже 2000 лет до н.э.». И далее: «Величина 3400 лет назад может рассматриваться как нижняя граница для времен экспансии Банту... Если мы возьмем наиболее частые гаплотипы среди тех 148 хромосом как предковые, и приложим к ним величину 0,00069, то время для М191 получится 14700 лет.... В этом случае, экспансия Банту могла произойти ранее, чем 3500 лет назад». И далее идут долгие рассуждения с неопределенной концовкой.
Если это «калибровка», то что такое НЕ калибровка?
К сожалению, исходные данные (гаплотипы) в статье не приведены, ссылка статьи, что они «в сети», не дает никакого линка. Я могу только заключить, что рассматриваемая популяция банту молодая, не более 1000—1200 лет до общего предка, как это часто бывает с африканскими популяциями, только недавно прошедшими очередное «бутылочное горлышко». Ни к каким археологическим или лингвистическим данным это не имеет никакого отношения. Рассуждения в статье не имеют никакого значения в отношении рассматриваемого вопроса. Никакой «калибровки» не было и близко.
В разделе «Материалы и методы» сообщается, что у авторов были 22 гаплотипа маори и 23 гаплотипа с островов Кука, гаплогруппы С2-М208, и эти данные действительно приведены в Таблице 1 в статье в виде серии 10-маркерных гаплотипов у маори, 10- и 7-маркерные гаплотипов у островитян Кука, и 10-маркерных гаплотипов у четырех самоанцев. Далее, сообщается, что маори прибыли в Новую Зеландию не позднее, чем 800 лет назад, что именно эта величина будет далее использована в статье (то есть 32 поколения назад, поскольку авторы брали 25 лет на поколение).
Применяя квадратичный метод, авторы показали, что среднее число мутаций на маркер у маори и островитян Кука равно 0,00998, при этом самоанцев в расчет не включали, а маори и островитян считали по 7-маркерным гаплотипам. По 10-маркерным расчета (у маори, или маори и самоанцев вместе) не было. На основании этого авторы посчитали, что скорость мутации равна 0,00998/32=0,000312.
Как видно, это вовсе не 0,00069, а даже в два раза медленнее. Далее авторы заключили, что это, скорее всего, недооценка, и опять пустились в долгие и вязкие рассуждения, почему это может быть недооценкой. На этом «калибровка» этой серии гаплотипов была завершена.
Авторы сообщили — со ссылками — что цыгане прибыли в Европу 900—1000 лет назад, и что они были зафиксированы в Болгарии ~700 лет назад (то есть ~28 поколений — А.К). Далее они сообщили, что анализировали 179 гаплотипов из 12 цыганских таборов в Болгарии, и что все гаплотипы имели снип М82. Далее, один гаплотип (единственный из табора Даракчи) был исключен, потому что оказался нечетным (? — А.К), и усреднение по всем 8-маркерным гаплотипам дало в среднем 0,01272 мутаций на маркер, и 0,01272/28=0,000454 как средняя скорость мутаций на маркер. Заметим, что это опять заметно ниже, чем якобы «калиброванная» 0,00069. Авторы опять стали обсуждать, что это должна быть «недооценка».
После этого авторы решили пересчитать данные, внеся коррективы в исходные серии гаплотипов, а именно внеся «веса» в гаплотипы полинезийцев, и сняв ряд гаплотипов цыган. Это привело авторов к «корректировке» средней скорости мутаций в серии полинезийцев от 0,000312 до 0,000705 (!), то есть более чем в два раза. Более того, с учетом погрешности таких поправок величина средней скорости мутации стала равна 0,000705±0,000332, со стандартным отклонением по маркерам 0,00078, то есть выше, чем сама скорость мутации.
В отношении цыган авторы решили, что один табор — «Музыканты» — очень «гетерогенный», потому что их гаплотипы отличаются на две аллели в шести гаплотипах из 19 от наиболее распространенного гаплотипа. Так что «Музыкантов» сняли со счета, все 19 гаплотипов, и опять ввели «веса» по размеру популяции (что совершенно нельзя делать в случае вполне однородной серии гаплотипов, и нельзя было делать в этом конкретном случае). Получили среднюю скорость мутации 0,000725±0,000187. То есть сблизили полученные величины для полинезийцев и цыган.
Если это «калибровка», тогда непонятно, что такое «калибровка» в понимании авторов.
Наконец, используя такие же подходы, а именно вводя «веса», авторы посчитали данные для 52 популяций по всему миру (! — А.К.), взяв данные из литературы, и получили для средней величины скорости мутации 0,000638±0,000109. Непонятно, как это может рассматриваться как «калибровка», потому что средний возраст 52 популяций в мире определить просто невозможно.
В статье это не пояснено, но описано в общих словах, что это делалось с привлечением аутосомальных данных для трех- и четырехнуклеотидных повторов, для которых скорости мутации равны, как полагают авторы, 0,00070 и 0,00071, соответственно. Конкретных данных не показано.
Это, конечно, никакая не калибровка. Это — подгонка.
А какие скорости мутаций на самом деле применимы для полинезийцев и цыган, если не изымать гаплотипы и не вводить никаких «весов»? Давайте определим. Иначе говоря, забудем на время о «методе Животовского» и посчитаем как надо.
У полинезийцев — маори (Новая Зеландия), жителей островов Кука, и самоанцев — в цитируемой статье суммарно было определено 37 10-маркерных гаплотипов. Дерево гаплотипов, построенное мной, приведено ниже.
Из дерева гаплотипов неясно, один предок или несколько. Проверим по критерию сходимости линейного и логарифмического методов.
Базовый гаплотип (в формате FTDNA плюс DYSA72, он же DYS461):
от которого все 37 гаплотипов имеют 49 мутаций. Константа скорости мутации для указанных 10-маркерных гаплотипов равна 0,018 мутаций на гаплотип на условное поколение (25 лет). Таким образом общий предок всех 37 гаплотипов жил 49/37/0,018=74→80 поколений то есть 2000±350 лет назад.
На дереве — 8 базовых гаплотипов (на вершине дерева). Это дает [ln(37/8)]/0,018=85→93 поколения, то есть 2325 лет. Это — в пределах погрешности расчетов. На самом деле разница в 16% между логарифмическим и линейным методами может означать небольшую примесь потомков еще одного общего предка в серии, но погрешность от этого, как мы видели, невелика.
Если же попытаться использовать «скорости Животовского» 0,00069 мутаций на маркер на поколение, то получится 49/37/10/0,00069=192 поколения до общего предка, то есть 4800 лет до общего предка рассматриваемой группы гаплотипов. Напомним, что авторы цитируемой статьи использовали время прибытия 800 лет назад. Потому и получили до смешного малую среднюю скорость мутации. Более того, авторы не понимают простого положения, что прибыть полинезийцы могли 800 лет назад, но это вовсе не означает, что это есть начало ДНК-генеалогической линии. Если они прибыли группой, то «принесли» общего предка популяции с собой. Иначе в США гаплотипы группы R1b все имели бы общего предка в 1620 году. Вот этот примитивизм мышления и интерпретации данных тоже несколько удивляет. Но удивляет не сильно, глядя на примеры, приведенные ранее в этой статье.
Вот такая цена «калибровки» Л. Животовским. Им была взята неверная величина, и по неверной «откалибровано».
Гаплотипы цыган были определены у 179 человек из двенадцати болгарских таборов (Zhivotovsky et al, 2004). На удивление, у всех гаплотипы похожи, явно от общего предка. Напрашивается объяснение, что в Средние века в Болгарию пришел очень ограниченный контингент цыган, может, всего один табор. Остальные, видимо, не прошли «бутылочное горлышко» популяции, и всего один патриарх дал выжившее потомство. Или патриархами были близкие родственники.
Рассмотрим гаплотипы цыган как в 6-маркерном, так и в 8-маркерном вариантах, чтобы сделать пример еще более наглядным, и проверить воспроизводимость нашего подхода. В цитированной статье были приведены 8-маркерные гаплотипы, но с произвольными удалениями, как показано выше.
В наиболее многочисленном таборе «Рудари» 62 человека из тестированных 67 имели следующий гаплотип:
Этот же гаплотип имели 12 из 13 человек табора «Кальдераш», 9 из 24 человек табора «Лом», все четверо тестированных из табора «Торговцы», 20 из 29 из табора «Калайджи», 12 из 19 из табора «Музыканты». В остальных гаплотипах мутаций тоже мало. Ясно, что гаплотипы «молодые», и им действительно всего несколько сотен лет.
Всего на 179 гаплотипов тестированных цыган пришлось 34 мутации, что дает 34/179/0,0088 = 22±4 поколений до общего предка, то есть 550±100 лет назад. Действительно, Средние века.
Сравним с расчетом по остаточным базовым гаплотипам. Их — 146 из всех 179. Это соответствует 23±3 поколению до общего предка (ln 179/146, деленное на 0,0088), или 575±75 лет назад. Практически, то же самое время.
Итак, и у цыган нормальная ДНК-генеалогия, при которой имеется весьма жесткое соответствие между числом убывающих базовых гаплотипов и накоплением мутаций в их производных. Не нужно было манипулировать с гаплотипами и удалять неугодные. Все гаплотипы там были на своих местах.
Из независимых источников, присутствие цыган в Болгарии было отмечено 700 лет назад (Zhivotovsky et al, 2004). Однако, эти сведения не обязательно точны, они могут относиться к другим цыганам, не обязательно гаплогруппы H1, потомство у первых цыган могло не выжить, и так далее. Тем не менее, 550—575 лет назад — это вполне удовлетворительное совпадение.
При переходе к 8-маркерным гаплотипам базовым гаплотипом стал следующий:
где последние два — маркеры DYS389-1 и 389-2. Они добавили во всех 179 гаплотипах 25 мутаций, то есть стало 59 мутаций. Это дает 59/179/0,013=25±4 поколений до общего предка, то есть 625±100 лет назад. Практически, то же самое, что и для 6-маркерных гаплотипов (34/179/0,0088=22±4 поколений до общего предка, то есть 550±100 лет назад).
В 8-маркерной серии — на 20 базовых гаплотипов меньше, чем в 6-маркерной серии, то есть 126. Получаем [ln(179/126)]/0,013=27±4 поколений до общего предка. Практически та же самая величина, 675±100 лет до общего предка.
Остается только гадать, как Л. Животовский получил примерно такую же величину (700 лет) при величине средней скорости мутации 0,000454, которая после некоторых манипуляций превратилась в 0,000725, а потом и в 0,00069.
Вот такая цена его «калибровкам».
Осталось с интересом перечитать слова Хаммера и самого Животовского в их последней статье (октябрь 2009): «При изучении вариаций в гаплотипах ...в популяциях с документированными непродолжительными хронологиями, они пришли к выводу об эволюционных эффективных скоростях мутации 0,00069 за 25 лет... (Zhivotovsky et al, 2004)... (то есть эти эффективные скорости были калиброваны за период времени примерно 1000 лет). С 2004 года этот подход был применен исследователями и критически рассмотрен в научной литературе» (например, Arredi et al. 2004; Di Giacomo et al. 2004: Gayden et al. 2007; Sengupta et al. 2006; Xue et al. 2005; Zhivotovsky and Underhill 2005).
Посмотрим, как, например, первый автор в этом списке «критически» рассматривал «метод Животовского». Они рассматривали популяции в Северной Африке, и решили рассчитать время до общего предка. Опять повторив мантру про «effective mutation rate calibrated by historical events (Zhivotovsky et al, 2004)» (мы сейчас знаем, что это была за «калибровка по историческим событиям»), они получили для гаплогруппы E3b2 величину 6900 лет до общего предка. Видимо, «критическое рассмотрение» состояло в том, что, не доверяя «методу Животовского», они пересчитали эти данные с помощью метода Кайзера для пар отец-сын, но, чтобы подтянуть данные к «методу Животовского», взяли для результатов «по Кайзеру» уже 30 лет на поколение. Получили 4200 лет до общего предка. Взяли бы 25 лет на поколение, как у Л. Животовского, получили бы 3500 лет для гаплогруппы E3b2, а не 6900 лет. В итоге авторы составили таблицу, в которой дали две колонки цифр — одну «по Животовскому», с поколением в 25 лет, другую «по Кайзеру», с поколением в 30 лет. «По-Животовскому» везде было в два раза выше. Так параллельно и обсуждали, не имея понятия, какие цифры им стоит обсуждать. К заключению о том, какие времена правильнее, так и не пришли. Видимо, это и есть «критическое рассмотрение».
Вот так рождаются басни про «калибровку» и «критическое рассмотрение». Хороший пример манипуляций и подтасовок в академической науке. Хороший пример того, чего нужно сторониться.
В итоге в этой части «академической науки» создалась некая эшелонированная оборона, в которой действует «циклический аргумент» о том, что якобы метод проверен и «калиброван», критически рассмотрен в литературе и таким образом доказан. Поскольку в этой «академической науке» задействованы авторитеты, то мало что понимающие в расчетах, «популяционные генетики» бездумно цитируют дефектный подход, что в свою очередь подается авторитетами как «критическое рассмотрение» и дополнительное обоснование метода. На самом деле не было ни калибровки, ни обоснования, ни критического разбора. Точнее, критический разбор был, и не один (см., например, Klyosov, 2008b, Клёсов, 2009е, Dienekes, 2008). Последний был озаглавлен «How YSTR variance accumulates: a comment on Zhivotovsky, Underhill and Feldman (2006)» ( http://dienekes.blogspot.com/2008/07/how-y-str-variance-accumulates-comment.html ).
Более того, этот подход уже давно служит предметом насмешек среди людей, понимающих в расчетах, например, на форуме RootsWeb, но «авторитеты» эти обсуждения игнорируют, продолжая говорить о «калибровке» и «критическом разборе». Мотив простой — эта критика не была опубликована в «академической печати».
Наконец, критика опубликована (Klyosov, 2009а). Ответ авторов «подхода» и тех, кто его применяет, последовал (Hammer et al, 2009b).
Что же в ответе? Восемь положений. Что же посчитали главным авторы ответа?
1. Оппонент (то есть я) выступил с критикой слишком быстро, что «не есть в интересах основательного научного процесса».
2. В свой критике я ссылаюсь на недоступные и неопубликованные работы (в печати в JoGG)
3. Использую нестандартную терминологию, как, например, «логарифмический» и «линейный» методы.
4. Я сделал неверный вывод, что гаплотипы образуют «генеалогию» и к ним могут применяться «генеалогические» подходы (то есть не «популяционные»).
5. Я не учитывал «генетический дрейф» или «никакие другие особенности мутаций в микросателлитах».
6. Авторы не знают, насколько мой метод применим к их системе.
7. Применяемый метод Животовского правильный, откалиброван и критически рассмотрен в научной печати.
8. Применяемые мной «деревья гаплотипов» являются интересными и могут быть в высшей степени полезными, но к ним нужно относиться с «большой осторожностью».
Положение (1), конечно, вызывает смех. Авторы, видимо, не в курсе, что «метод Животовского» уже давно вызывает насмешки, что я его давно и детально разобрал, и на это описание не нужно времени.
Положение (2) можно было бы принять, если бы не русскоязычные Л. Животовский и Т. Карафет в авторах. Один вопрос мне в частной переписке, если они не умеют пользоваться Интернетом, и я бы дал им ссылки на работы, на которые они легко могли бы взглянуть. Мог бы и выслать. Если бы они хотели. Но желания, явно, не было.
Положение (3) — авторы просто не владеют предметом. Логарифмический метод в химической кинетике существует уже десятки лет. Он подробно и в азбучном варианте изложен в учебнике И.В. Березина и А.А. Клёсова «Практический курс химической и ферментативной кинетики», который уже более 30 лет служит основным учебником по данному предмету для университетов. Опять — короткий вопрос со стороны ЛЖ или ТК, и они узнали бы для себя много нового. Но не захотели.
Положение (4) — авторы просто не поняли, о чем речь. А речь о том, что дерево гаплотипов можно разложить по ветвям, и к каждой ветви приложить «генеалогический подход» и «генеалогическую скорость мутаций». А вот обрабатывать все дерево чохом, как делают авторы, в общем случае неверно.
Положение (5) — авторы опять не поняли, что разложение дерева по ветвям — это и есть учет «генетического дрейфа».
Положение (6) — в моем «Комментарии» все описано. Авторы просто не захотели, или не смогли разобраться.
Положение (7) — неверно и подтасовочно. В настоящей статье я это еще более подробно описал.
Положение (8) — наконец-то разумные слова. Следует, правда отметить, что к «методу Животовского» слова про «осторожность», по мнению авторов, видимо, неприменимы. Там осторожность не нужна. Это только к другим авторам относится.
Воистину, двойной стандарт еще и забавен.
Хочу еще раз подчеркнуть, что вклад авторов в биологическую сторону вопроса сомнению не подлежит. Но когда авторы полагают, что они знают, как вести расчеты динамики мутаций в гаплотипах, что определенно НЕ их сильная сторона, когда занимаются подтасовками, искажениями и преувеличениями, то мое уважение к ним сильно падает. Хотя авторам есть оправдание — их фактически ввел в заблуждение метод Л. Животовского, которому они просто доверились. Вред, который последний нанес ДНК-генеалогии свои «методом», еще будет оценен, и будет определенно оценен как вред немалый.
В рассматриваемой работе автор, Е.Я. Тетушкин, сетует, что калибровка скоростей мутаций, основанная на археологических «и прочих непрямых свидетельствах» часто «очень неточна и ненадежна», а вот прямые результаты скоростей мутаций по парам отец-сын якобы намного точнее, если только убедиться, что нет проблемы с отцовством. Тогда можно просто разделить число наблюдаемых мутаций на число мейозов. И в качестве примера автор приводит недавнюю работу Ballantyne et al (2010) по установлению скоростей мутаций для 186 маркеров почти в 2000 пар отец-сын.
Этим «теоретикам» в популяционной генетике, которые сами не считают, но думают, что все так просто и надежно, можно только порекомендовать опробовать свои рекомендации самим. На самом деле то, что приводится в статье Ballantyne et al (2010) и других подобных статьях, совершенно неприменимо для реальных расчетов в ДНК-генеалогии. Может в судебной практике это и полезно, но не при расчетах времен жизни общих предков.
На первый взгляд кажется, что в той статье — замечательная статистика, и вот наконец-то получены данные, которые позволят откорректировать скорости мутаций для протяженных гаплотипов и вывести ДНК-генеалогию на новый количественный уровень. Закончились споры и дискуссии, какие величины скоростей мутации самые правильные, и осталось только щелкать древние (и современные) гаплотипы как орехи. Видимо, этой эйфорией прониклись и сами авторы, поскольку в абстракте статьи написано черным по белому, после фразы, что теперь можно анализировать как близких, так и удаленных родственников «This finding is expected to revolutionize Y-chromosomal applications...»
Увы, не тут-то было.
Во-первых, поскольку мутации в гаплотипах обычно редки, то этих почти двух тысяч пар отец-сын явно недостаточно для хорошей статистики. Во многих маркерах мутаций не было вообще, в большинстве маркеров по одной-две-три мутации на все две тысячи пар, что вовсе немного для статистики. Понятно, что если мутаций в маркере вообще нет, то он остается неохарактеризованным. Одна мутация означает статистическую погрешность плюс-минус 100%, поскольку где одна мутация, то там легко могло быть две или ни одной. Это можно показать и количественно, в терминах математической статистики. Две мутации — это погрешность примерно плюс-минус 70%. Ясно, что о расчетах на основе отдельных маркеров здесь не может быть и речи. Не случайно в предыдущих статьях на эту тему я писал, что для статистически надежных данных нужны серии из сотен тысяч, а то и миллионов пар отец-сын, что практически нереально, пока, во всяком случае.
Во-вторых, для каждого маркера изучалось разное количество пар отец-сын. Это почти неизбежная проблема в таких массовых исследованиях, поскольку часто имеют место сбои, аллели не определяются, лаборанты ошибаются, а переделывать — дорого и неохота. Скорее второе, чем первое. В итоге итоговая таблица данных выглядит как лоскутное одеяло, с массой прорех. Но это самая незначительная проблема, поскольку данные можно пересчитать на некую «нормированную» величину, что мы ниже и сделаем двумя способами.
В-третьих, авторы не ориентировались на ДНК-генеалогию, а преследовали свои цели. В итоге ряд важных маркеров определен вообще не был, а именно из 37- и 67-маркерной панелей. Поэтому для этих расчетов, с использованием наиболее ценных, протяженных гаплотипов, данные обсуждаемой статьи вообще непригодны.
В-четвертых, и это очень важно, данные в парах отец-сын имеют принципиальный, системный недостаток в рамках понятий ДНК-генеалогии. Они позволяют рассчитать скорости мутации только НА ПОКОЛЕНИЕ, просто по определению. А исторические расчеты, понятия, исследования ведутся в годах. Переход же от числа поколений к числу лет принципиально невозможен со сколько-нибудь приемлемой точностью. Этот «коэффициент» — величина плавающая, и меняется от эпохи к эпохе, от культуры к культуре, и вообще зависит от массы факторов. В разных работах популяционных генетиков он варьируется от 18 до 35 (как минимум) лет. Соответственно, при этих величинах число лет будет варьироваться почти в два раза, и вся точность определения скоростей мутаций в парах отец-сын сводится на нет. Один выход из ситуации — калибровать полученные данные по историческим и генеалогическим данным, что мы и так делаем без тех хлопот.
Это — к «революционированию» науки, о чем в состоянии эйфории воскликнули авторы статьи. Как видно, революционирования особого не видно, хотя исследование полезное, как мы ниже покажем. Вся его полезность в основном сводится к тому, что показывает, что наши значения скоростей мутации, определенные эмпирически, вполне согласуются — по порядку (!) — с расчетами по парам отец-сын.
Напомним сначала основные положения нашего эмпирического подхода и соответствующие величины констант скоростей мутации для разных протяженных гаплотипов, а потом посмотрим, что в этом отношении дают результаты рассматриваемой статьи.
Принципиальная методология нашего подхода описана во многих статьях. Число мутаций в сериях гаплотипов может рассматриваться с применением любого из четырех основных методов: «линейный», «логарифмический», «квадратичный» и «пермутационный». Самый простой — логарифмический, в котором и мутации считать не нужно, считаются только немутированные гаплотипы, и берется логарифм отношения числа всех гаплотипов в серии к числу немутированных гаплотипов и далее к средней скорости мутации маркеров в гаплотипе. Но перед этим необходимо убедиться, что вся рассматриваемая серия гаплотипов происходит от одного общего предка. Это, впрочем, непременное требование ко всем четырем методам расчетов. На это есть критерии, которые применяются к дереву гаплотипов рассматриваемой серии. Для линейного метода необходимо определить среднее число мутаций на маркер в данном гаплотипе, разделить на среднюю скорость мутаций и сделать поправку на возвратные мутации. Такую поправку необходимо делать и в логарифмическом методе. В квадратичном и пермутационном методах поправки на возвратные мутации делать не нужно, но эти два метода особенно чувствительны к примесям гаплотипов от других общих предков.
В любом методе необходимо использовать среднюю скорость мутации — на маркер или на гаплотип:
Для 12-маркерных гаплотипов — 0,020 мутаций на гаплотип, или 0,00167 мутаций на маркер,
Для 25-маркерных гаплотипов — 0,046 мутаций на гаплотип, или 0,00183 мутаций на маркер,
Для 37-маркерных гаплотипов — 0,090 мутаций на гаплотип, или 0,00243 мутаций на маркер,
Для 67-маркерных гаплотипов — 0,12 мутаций на гаплотип, или 0,00179 мутаций на маркер,
Для 111-маркерных гаплотипов — 0,198 мутаций на гаплотип, или 0,00178 мутаций на маркер.
Эти скорости мутаций откалиброваны при величине 25 лет на поколение. Последнее — фиксированная математическая величина, а не какое-либо иное соображение. Дело в том, что при расчетах в ДНК-генеалогии, исходя из числа мутаций или числа немутированных гаплотипов в серии, всегда получается величина <kt> , где k — это средняя скорость мутаций (или константа скорости мутаций), и t — число поколений до общего предка серии гаплотипов. Например, ранняя серия данных для Клана МакДоналдов гаплогруппы R1a («Красная подгруппа» в номенклатуре Клана) содержала 44 мутации в 68 12-маркерных гаплотипах, и в последнем варианте (сентябрь 2011 года) было 81 мутации в 143 12-маркерных гаплотипах. Поскольку мы знаем, что общий предок всей группы, Джон, Лорд Островов, жил (в контексте данного исследования) 650 лет назад (он умер в 1386 году, то есть 624 года назад), то мы считаем это как 26 поколений по 25 лет каждое. Иначе говоря, мы калибруем константы скорости мутаций при заданной математической величине в 25 лет на поколение. Мы могли бы положить это как 13 поколений при 50 годах на поколение, и получили бы тот же результат, а именно 650 лет до общего предка. Просто скорость мутации стала бы вдвое выше.
Давайте посмотрим, что получается. Задавая 26 поколений до общего предка (при 25 годах на поколение) получаем 12-маркерные гаплотипы, для ранней и последней серий гаплотипов:
44/68/26=0,025±0,004 мутаций на гаплотип на поколение,
81/143/26=0,022±0,002 мутаций на гаплотип на поколение.
Из 143 гаплотипов 86 были одинаковыми, то есть базовыми. Применяя логарифмический метод, получаем: [ln(143/86)]/26=0,020±0,002 мутаций на гаплотип на поколение.
Видно, что эти величины равны в пределах погрешности. Более детальные последующие исследования со многими сериями гаплотипов позволили уточнить константу скорости мутации как 0,020± 0,001 мутаций на гаплотип на поколение.
Таким же образом калибруются и более протяженные панели гаплотипов, и не только по Клану МакДоналдов, а по многим документальным генеалогиям. Это детально описано в недавней статье в англоязычном журнале «Успехи антропологии».
В то же время данные Ballantyne et al для индивидуальных маркеров не могут быть использованы для расчетов, поскольку статистики там практически нет. Так, для числа мутаций по почти 2000 пар отец-сын для первых 12 маркеров, а именно 3, 2, 7, 5, 3, 6, 0, 0, 6, 9, 1, 6, статистические погрешности, или доверительные интервалы соответственно равны (при 95% надежности) соответственно плюс-минус 115%, 141%, 76%, 89%, 115%, 82%, данных нет, данных нет, 82%, 67%, 200%, 82%. Ясно, что с такими погрешностями в расчетах делать нечего.
Расчеты показывают, что для того, чтобы пары отец-сын дали статистически значимые константы скорости мутации для индивидуальных маркеров, должно наблюдаться не менее 400 мутаций в каждом локусе (это даст погрешность ±10% при 95%-й надежности расчетов). Это условие будет выполняться для минимум 800 тысяч (!) пар отец-сын, и даже при этом наиболее медленные маркеры дадут всего одну или несколько мутаций на локус. Но даже и в этом случае полученные константы скорости мутации будут относиться всего к одному поколению, и не будут применимы для расчетов TMRCA в годах, а в исторических науках в поколениях не считают. Так что опять придется подгонять к уже известным величинам констант скоростей мутаций. Круг замкнулся.
Вот такая популяционная генетика с ее надеждами, что пары отец-сын — это то, что нужно для повышения надежности расчетов. «Страшно далеки они от народа», эти популяционные генетики.
Это стало традицией, и уже не печальной, а рутинной. Появляется очередная статья «титулованных» (или начинающих, разницы нет) популяционных генетиков, иногда с примкнувшими к ним лингвистами (как в данном случае), которые тоже ничего не понимают в ДНК-генеалогии, и уже ясно, что толк от статьи будет только в виде приложения со списком гаплотипов. Саму статью можно не читать, чтобы не расстраиваться.
А расстраиваться есть чему — уже который год эти и другие попгенетики производят совершенный мусор, другого названия их продукции нет (кроме списка гаплотипов, да и то в этом случае, как часто бывает, авторы выбросили из него два локуса, потому что не умеют с ними работать. В итоге из 19-маркерных гаплотипов в списке оставили 17-маркерные, но и на том спасибо). Поскольку авторы, как обычно, применили «метод Животовского» для расчета датировок, то, как обычно, получили сапоги всмятку, с ошибками датировок примерно между лишними 100% и 300%, то есть ошиблись в 2—4 раза. Если бы ошибка была систематической, то есть одной и той же, то еще куда ни шло, можно было бы внести стандартную поправку. Но с этой «популяционной константой Животовского» ошибка всегда разная и зависит от того, какие именно гаплотипы, насколько древний предок, какое соотношение между ветвями в популяции, то есть их относительная численность, и так далее. Это все варьируется от серии к серии гаплотипов, поэтому получаемые данные не только завышенные, но вообще не имеют никакого смысла.
Так, для гаплогруппы R1a в Индии авторы получают датировки общих предков от 10 до 15 тыс. лет назад, и, понятное дело, тут же утверждают, что это не арии, потому что арии в Индию «по некоторым данным» пришли только «примерно 3 тыс. лет назад». На самом деле мы понимаем, что по «популяционным скоростям» это как раз могут быть арии, но поскольку без специального анализа их списка гаплотипов это утверждать нельзя, то утверждать по их данным вообще ничего нельзя.
Авторы, однако, уверовав, что это не арии, пишут, что по датировкам это должны быть «автохтоны», а поскольку они, эти гаплотипы, принадлежат отчасти высшим кастам, то авторы уже пишут, что касты были созданы много ранее прибытия ариев, которые, судя по полученным данным, в Индию вообще не приходили. И начинаются долгие и бессмысленные рассуждения об истории Индии, о временах «социальной стратификации» на основе полученных данных, о временах продвижения туда сельского хозяйства, и о том, что полученные датировки хорошо коррелируют с этими временами. На самом деле там НИЧЕГО НИ С ЧЕМ не коррелирует, потому что результаты статьи неверные и лишенные смысла. То есть если называть вещи своими именами — это подтасовки.
Что забавно — это то, как авторы на многих страницах тщательно описывают математическую и статистическую обработку данных, как ведут расчеты с точностью «до 95%», как гоняют гаплотипы по программам по 5 тысяч повторов, и методом Монте-Карло по 500 тысяч раз... Но это все — опять же бессмысленно, поскольку сама методология расчетов неверна. Расчеты «разнообразий» (diversity) гаплотипов, столь любимые попгенетиками, тоже практически ни для чего в статье не используются. Что толку сообщать о том, что «разнообразие» среди всех популяций равно 0,886, среди племен 0,796, среди каст 0,881? После этой «информации», авторы к этим цифрам и не возвращаются. Это у них ритуал такой. А поскольку в этих «разнообразиях» перемешаны все гаплогруппы и их ветви, и старые и молодые, и разной численности, то смысла в них действительно никакого нет.
Авторы решили, что если гаплогрупп много и «разнообразие» высокое — то это «автохтоны», которые появились в Индии в позднем плейстоцене (30—10 тыс. лет назад) — в такие попала, в частности, и гаплогруппа R1a. А те, которых меньше по численности и которые помоложе — то, значит, пришельцы из Западной Евразии в ходе последних 10 тыс. лет, в число которых попали J2, E, G, L3. Понятие «бутылочных горлышек» популяции авторам, видимо, неведомо. Или они его своеобразно понимают, хотя жонглируют словами «дрейф», «эффект основателя» и подобными. Хотя дальше авторы пишут, что ВСЕ гаплогруппы в Индии имеют возраст свыше 10 тыс. лет. Это понятно, поскольку с помощью «метода Животовского» завышают возраст всех гаплогрупп в 3—4 раза. Естественно, рассчитывают опять же любимые индексы Fst, опять, естественно, не разделяя гаплогрупп.
Поэтому читать статью действительно не стоит. Кроме, пожалуй, того, чтобы узнать, откуда взялись гаплотипы в Приложении к статье. А затем взять эти гаплотипы и провести свой анализ. Чтобы от той статьи был хоть какой-то прок.
Мы и провели свой анализ всех 1680 17-маркерных гаплотипов, собранные в 12 племенах и 19 кастах крайнего юга Индии, штата Тамил Наду. Это — крайняя юго-восточная провинция Индии. Жители племен были из удаленных деревень тропических лесов, члены каст тестировались, в частности, в колледжах. Брамины принадлежали к четырем популяциям, все «индоевропейские». Одна относительно небольшая по численности, со «средним» социальным рангом (язык — саураштри), три остальные — «высокого» социального ранга, их язык по рождению — санскрит. 82% гаплотипов, по данным авторов, относятся к семи гаплогруппам — Н1-М52 (17,4%), F*-M89 (16,3%), L1-M27 (14,0%), R1a1-M17 (12,7%), J2-M172 (9,4%), R2-M124 (8,2%), Н-М69 (4,7%). Остальные, минорные, относились к гаплогруппам С-М130, Е-М96, G-M201, Н1а-М197, J-M304, J2al-M47, J2a3-M68, К-М9, L3-M357, O-М175, Р-М45, R-M207.
Не буду утруждать читателя детальным разбором данных, это все опубликовано. Ссылки есть в прилагаемом списке литературы.
А что вообще ожидалось найти? Ожидаем ли мы получить что-то новое и необычное? Ведь индийские гаплотипы гаплогруппы R1a мы рассматривали немало раз, все это было опубликовано. Мы знаем, что в Индии есть два «типа» гаплотипов группы R1a, одна «автохтонные» (пришли, скорее всего, с востока, со стороны Алтая и/или Северного Китая примерно 12—13 тыс. лет назад, от них остались ветви с общим предком давности 7—8 тыс. лет), другие «индоевропейские», арии, прибывшие в Северную Индию примерно 3500 лет назад. Последние по своим базовым гаплотипам близки к таковым на Русской равнине, и в 67-маркерном формате имеют вид
или
или
Первый базовый гаплотип относится к субкладу R1a-Z280 (центральный евразийский), общий предок которого жил 4900±500 лет назад, второй — R1a-Z93-L342.2 (юго-восточный евразийский), с общим предком, жившим в те же времена, 4900±500 лет назад, третий — R1a-Z93-L342.2-L657, с общим предком примерно 4400±470 лет назад (см. ниже). Как видно, базовые гаплотипы их почти идентичны. Более детальный анализ показывает, что их общий предок относился к евразийской ветви Z283 и жил примерно 5500 лет назад, видимо, в Европе. От него разошлись и центрально-европейская ветвь, и скандинавская, и центрально-азиатская, и южно-восточная, и арийская (степная) ветвь, как подветвь южно-восточной ветви, и далее подветвь L657.

Естественно, этих сведений не было в обсуждаемой статье (ArunKumar et al, 2012). Не было там ни предковых гаплотипов, ни ожидаемых датировок, ни картины субкладов, вообще ничего, что представляет интерес для ДНК-генеалогии.
По состоянию на начало декабря 2012 в нашей базе данных IRAKAZ (где первые буквы означают Игорь Рожанский, Анатолий Клёсов, Александр Золотарев) числились 34 67-маркерных гаплотипа R1a-L657, из которых 24 были арабскими (Саудовская Аравия, ОАЭ, Кувейт, Бахрейн, Катар), пять гаплотипов были из Индии, три из Пакистана и два из Казахстана. Все 34 гаплотипа содержали 613 мутаций от приведенного выше базового гаплотипа, что дало 613/34/0,12=150→176 условных поколений, то есть 4400±470 лет до общего предка субклада L657.
Если рассмотреть десять гаплотипов только Индии, Пакистана и Казахстана, то они дают точно такой же базовый гаплотип, как и все 34 гаплотипа группы L567 (см. выше), и содержат 163 мутации, что дает 163/10/0,12=136→158 поколений, или 3950±500 лет до общего предка. В принципе, это в пределах погрешности расчетов времени жизни общего предка для всех 34 гаплотипов, но в любом случае ясно, что общий предок у них был один или близкородственный.
Итак, мы можем ожидать, что среди индийских гаплотипов мы найдем линии L342.2 и/или L342.2-L657, общие предки которых жили 4900—4400 лет назад, или более недавние, если арии прошли «бутылочное горлышко» популяции в ходе прибытия в Индию или в ее южную часть, а также автохтонные линии с «возрастом» намного больше, с гаплотипами, содержащими необычные (для «индоевропейских», или арийских гаплотипов) мутации, как, например, DYS392=13 или 14, или сочетание DYS389=14-18 (общий предок которых для 46 гаплотипов Южной Индии жил 7200±960 лет назад. Помимо этого, мы ожидаем найти, что линии гаплогрупп G, J2 и других в Индии будут намного старше R1a. Это — не арийские гаплогруппы. Мы не ожидаем найти в Индии гаплогруппу R1b, что, впрочем, уже показано выше. Это тоже не арийская гаплогруппа. Эрбины (носители R1b) перешли на индоевропейские языки уже в Европе, намного позже времен прибытия ариев в Индию.
Так это все и оказалось при анализе данных.
Из всех 71 гаплотипов только две аллели в DYS389-1 были не равны 13. Это говорит о том, насколько четко разделились подветви. Во всех 71 гаплотипах было 248 мутаций от указанного базового гаплотипа. Это дает 248/71/0,030=116→132 поколения, или 3300±380 лет до общего предка ветви. Это в пределах погрешности совпадает с известным временем перехода ариев в Индию.
Обратим внимание на сходство выявленного базового гаплотипа описанной индийской ветви с базовым гаплотипом субклада L657, приведенным здесь для сравнения в 37-маркерном формате (см. выше):
Совпадение имеет место по всем маркерам (G АТАН4=12,6 в индийской серии, что округлено до 13 в показанном выше базовом гаплотипе, и DYS635 отсутствует в 67-маркерной панели FTDNA). Таким образом, эта подветвь с хорошей вероятностью представляет субклад R1a-L657. Надо отметить, что эта подветвь наиболее представительна в отношении браминов, их в составе ветви 22 из 35 браминов в гаплогруппе R1a.
Следующей за ней (в нижней части ветви на рис. 2 ( ?? ) ) идет подветвь с DYS389-1=14, в ней 68 гаплотипов. Базовый гаплотип:
Из всех 68 гаплотипов только одна аллель в DYS389-1 была не равна 14. Это опять говорит о том, насколько четко разделились подветви. Это, конечно, не только из-за одной аллели. Как видно из приведенного базового гаплотипа, различия между базовыми гаплотипами с DYS389-1=13 и DYS389-1=14 составляют пять мутаций (отмечены). На самом деле разница там меньше, суммарно 3,04 мутации, поскольку почти все мутации дробные.
Во всех 68 гаплотипах было 255 мутаций от указанного базового гаплотипа. Это дает 255/68/0,030= 125→143 поколения, или 3575±420 лет до общего предка ветви. Это опять совпадает с известным временем перехода ариев в Индию. Таким образом, в Индию вошли как минимум два арийских «племени», различающиеся своими общими предками. Разумеется, это вовсе не подразумевает, что они входили раздельно. Это могли быть, например, представители субкладов L657 и L342.2.
Поскольку их базовые гаплотипы различаются на 3.04 мутации, то есть на 3.04/0,030=101→112 поколений, или 2800 лет, то общий предок этих двух племен жил (2800+3300+3575)/2=4800 лет назад. Это действительно с хорошей вероятностью субклад R1a-L342.2 и/или L342.2-L657, хотя приведенный выше 17-маркерный базовый гаплотип несколько отклоняется от их базовых. Впрочем, всегда есть возможность того, что это новый, пока не описанный субклад юго-восточной ветви.
Следующей идут две подветви (на рис. 3 ( ?? )), верхняя с DYS389-1=14, их 15 гаплотипов, нижняя с DYS389-1=13, их 23 гаплотипа. Базовый гаплотип верхней:
Во всех 15 гаплотипах все аллели в DYS389-1 равны 14. Это опять говорит о том, насколько четко разделились подветви. Опять, это, конечно, не только из-за одной аллели. Как видно из приведенного базового гаплотипа, различия между двумя базовыми гаплотипами с DYS389-1=14 составляют четыре мутации (отмечены). На самом деле разница там меньше, суммарно 2,89 мутации, поскольку почти все мутации дробные. Во всех 15 гаплотипах было 46 мутаций от указанного базового гаплотипа. Это дает 46/15/0,030=102→114 поколения, или 2850±510 лет до общего предка ветви. Это уже несколько позже известного времени перехода ариев в Индию, и, возможно, эта ветвь уже «индоариев» в буквальном смысле слова (примерно первая половина 1 тыс. до н.э.). То, что она «молодая», говорит и то, что восемь маркеров в ней не мутированы, то есть одни и те же во всех 15 гаплотипах, или имеют всего одну мутацию.
Поскольку базовые гаплотипы обеих подветвей с DYS389-1=14 различаются на 2,89 мутации, то есть на 2,89/0,030=96→107 поколений, или 2675 лет, то общий предок этих двух племен жил примерно (2675+2850+3575)/2=4550 лет назад. Это — типичные времена для субкладов L342.2 и L657, Русская равнина.
Базовый гаплотип подветви из 23 гаплотипов:
Только три аллели в DYS389-1 здесь не равны 13. Опять, это, конечно, данная подветвь отделилась не только из-за одной аллели. Как видно из приведенного базового гаплотипа, различия между двумя базовыми гаплотипами с DYS389-1=13 составляют шесть мутаций (отмечены). На самом деле разница там меньше, суммарно 2,61 мутации, поскольку почти все мутации дробные. Во всех 23 гаплотипах было 83 мутации от указанного базового гаплотипа. Это дает 83/23/0,030=120→136 поколений, или 3400±505 лет до общего предка ветви, то есть опять известное время перехода ариев в Индию (середина 2 тыс. до н.э.). О том, что базовый гаплотип не очень «молодой», говорит и то, что только четыре самых «медленных» маркеров в ней не мутированы.
Поскольку базовые гаплотипы обеих подветвей с DYS389-1=13 различаются на 2,61 мутации, то есть на 2,61/0,030=87→96 поколений, или 2400 лет, то общий предок этих двух племен жил примерно (2400+3400+3300)/2=4550 лет назад, то есть та же величина, что и для времени жизни общего предка двух DYS389-1=14 ветвей. Это — типичные времена для субкладов L342.2 и L657, Русская равнина.
Подводя итог, заметим, что по полученным данным большинство южноиндийских гаплотипов группы R1a (177 из 196, представленных в списке, то есть 90%), берут свое начало в субкладе R1a-L342.2, образованном примерно 4900 лет назад, видимо, в Европе, и далее в последующем субкладе L657. На Русской равнине и далее по ходу миграции ариев этот субклад разделился на четыре ветви, общий предок которых жил примерно 4550 лет назад, и эти четыре ветви прибыли в Индию примерно 3600 лет назад. Их потомки живут в Индии и сейчас. Эти ветви дают довольно сложную суперпозицию аллелей, разделить которую на ветви было трудно, поскольку протяженных индийских гаплотипов пока мало. Почти 200 17-маркерных гаплотипов сделали такое разделение возможным.
Интересно, что ни башкирских, ни киргизских ветвей субклада L342.2 в Южной Индии не было. Для справки, их 17-маркерные базовые гаплотипы следующие:
Понятно, что речь здесь ни о башкирах или киргизах в Индии как таковых, и не о приходе их как этносов в Индию. Речь о том, что носители субклада L342.2, которые передали свои Y-хромосомы будущим киргизам и башкирам, не появились в Индии. Возможно, эти племена (или популяции, в более общей формулировке) там, с будущими киргизами и башкирами, и остались. Но субклад L342.2 — тот же.
Выше приведены четыре выявленные базовые гаплотипы индийских R1a субкладов. Только последний относительно близок к башкирским и киргизским гаплотипам (времена жизни общего предка 1125±190 и 2100±250 лет, соответственно), и он принадлежит общему предку, который жил 3400±505 лет назад, действительно во времена прихода ариев в Индию. Поскольку его базовый гаплотип расходится с башкирским и киргизским на 5,00 (часть мутаций дробные) и 4,57 мутаций (отмечено), соответственно, то есть на 5/0,03=167→200 поколений, или 5000 лет (башкиры) и 4.57/0,03=152→179 поколений, или 4475 лет (киргизы), соответственно, то общий предок индийского арийского гаплотипа и башкир или киргизов жил, соответственно, (5000+1125+3400)/2=4800 и (4475+2100+3400)/2=5000 лет назад. Это, разумеется, примерные оценки, но они позволяют предположить, что общие предки индийских, башкирских и киргизских гаплотипов восходят к началу субклада L342.2.
Остальные 19 гаплотипов группы R1a имели необычные мутации, и не принадлежали описанным выше ветвям. Они были рассеяны по всему дереву гаплотипов, попадая зачастую не в свои гаплогруппы. Некоторые из них имели явные ошибки в типировании (например, DYS392=7 вместо обычных 11, и тому подобные), или если это не ошибки — то это совершенно архаичные гаплотипы. Они не были включены в расчеты, но были приняты к сведению как возможные указания на наличие архаичных гаплотипов с списке южноиндийских R1a. В итоге 15 этих «рассеянных по дереву» гаплотипов с необычными мутациями были сведены в одну группу, в которой необычные (видимо, архаические) мутации включали серию из DYS390=22, DYS392=13 или 14, DYS439=13, и так далее. Все 15 гаплотипов содержали 182 мутации от условного базового гаплотипа, что дает 182/15/0,030=404→656 условных поколений, или 16400 лет до общего предка. Понятно, что о точности расчетов здесь говорить не приходится, поскольку эти 15 гаплотипов представляют собой разрозненные древние ветви. Тем не менее, полученная условная датировка в целом не противоречит датировке серии северокитайских гаплотипов группы R1a с возрастом общего предка около 20 тысяч лет.
К сожалению, геномный анализ во многом пошел по печальному пути популяционной генетики человека. Схема такая: с помощью самых передовых методов генетического анализа фиксируется картина мутаций в геноме определенной популяции, и затем совершенно безграмотно и бестолково проводятся его интерпретации.
Статья, упомянутая в заголовке (Elhaik, 2012), тому показательный пример. Автор задался целью выявить «хазарский след» и геномную связь его с европейскими евреями, ашкенази. Для этого он исходил из двух совершенно бестолковых положений. Первое — что хазары предположительно жили на Кавказе, и поэтому у них должен быть кавказский геном. Второе — поскольку они были евреями и в итоге передвинулись в Европу, то этот кавказский геном передвинулся с ними в Европу. А поэтому, если геном кавказский выявляется в геноме ашкинази, то это — хазарский след, и это является доказательством исходного положения.
Более бестолкового положения просто нельзя придумать. Дело в том, что половина Восточного и Северо-восточного Кавказа имеют гаплогруппы J1 и J2, которые к евреям не имеют прямого отношения, кроме того, что это одни и те же древние рода, что и у евреев. Они разошлись от одних общих предков в незапамятные времена, возможно, 10—15 тыс. лет назад. Это — не еврейские ДНК-линии. Но род — один, и, естественно, в геноме он дает те же самые многие мутации, что и у евреев. Хазары здесь совершенно ни при чем. Были они или не были — это не имеет ни малейшего отношения к тому, что геномы многих кавказцев и евреев схожи, и ведут начало своей схожести, как уже сказано, в доеврейские времена.
Эту схожесть автор рассматриваемой статьи, естественно, и нашел, и 90% его статьи длинно и нудно, со многими повторами повествует о геноме якобы хазар, в каком направлении якобы шли их якобы миграции, и что якобы открытия автора (подчеркивается, что впервые найдены доказательство наличия генома хазар) показывают, что евреи-ашкенази имеют не европейское происхождение (рейнская гипотеза), а именно хазарское (хазарская гипотеза), и что именно это в статье выявлено.
Читать статью досадно и неловко, неспособность автора даже минимально анализировать альтернативные варианты просто угнетает. Неужели так трудно сообразить, что сходство геномов кавказцев и евреев может иметь другие причины, чем через хазар? Неужели так трудно взглянуть на гаплогруппы кавказцев, и сравнить их с гаплогруппами евреев? А ведь там все основные гаплогруппы ашкенази — Jl, J2, G2, R1b, только у каждой своя история, отличная от истории евреев.
Вот, например, доля гаплогрупп J1 и J2 у восточных и северо-восточных кавказцев:
| Авары | J1 58% | |
| Чеченцы (без Ингушетии и Дагестана) | J2 58%, | J1 23% |
| Чеченцы (Дагестан) | J2 58%, | J1 15% |
| Чеченцы (Ингушетия) | J2 51%, | J1 21% |
| Даргинцы | J1 69% | |
| Ингуши | J2 82%, | J1 7% |
| Кайтаки | J1 85% | |
| Кубачинцы | J1 98% | |
| Лезгины | J1 44% |
Сплошные J1 или J2, или и то и другое. А у западных кавказцев — сплошные G2, которых у ашкенази опять много.
Вот это непонимание, что гаплогруппы Y-хромосомы дают важный вклад в геномный анализ, приводит к постоянным недоразумениям у генетиков. Это априори трудно было предсказать, но так получается. Недавно у генетиков опять был конфуз по этой части, когда они объявили, что у европейцев есть заметный процент генома «американских индейцев». Причем и у западноевропейцев, и у восточноевропейцев. У русских, например, в геноме тоже заметный вклад «американских индейцев». И никто не сообразил, что это абсурд. Так и продолжали повторять, как попугаи, продолжают до сих пор. И никто не сообразил, что это — гаплогруппа P, из которой вышли как гаплогруппа Q, основная у американских индейцев, так и гаплогруппы R1a и R1b, основные у восточноевропейцев и западноевропейцев, соответственно. Поэтому картина мутаций в геноме пересекается у всех трех указанных популяций.
Иначе говоря, геномный анализ дает правильные картины, но интерпретация попгенетиков никуда не годится. Типичная ментальность попгенетиков — видят это у современных америндов, видят это у европейцев — значит у европейцев это компонент американских индейцев. То, что это может быть и наоборот (что тоже было бы неверно), и это у америндов «европейская компонента», им в голову не приходит. А уж то, что это от общего предка — это для тех генетиков вообще уму непостижимо. Попгенетики мыслят только категориями современности, типа «что вижу, о том и пою».
Вот так же и с кавказским и еврейским геномом. Подумать о том, что это от древнего общего предка, для попгенетиков слишком сложно.
Эту цитируемую статью стоит переписать заново, и о хазарах ни слова не упоминать. Была бы хорошая статья. О том, как одинаковая гаплогруппа в значительной степени определяет картину генома; о том, что порой и геном не обязательно определять, гаплогруппа во многом рулит картиной генома. О том, что евреи и кавказцы — древние родственники, хотя это ясно и из ДНК-генеалогии. Хазары там просто под раздачу попали. Короче, статья в отношении хазар неверная, и о хазарах вопрос совершенно не решает. Но материал поучительный, как НЕ НАДО ставить эксперименты, если вопрос с самого начала неправильный.
Вот давайте под этим углом на материал и посмотрим.
В статье были рассмотрены данные для 1287 человек из 8 еврейских и 74 нееврейских популяций, которые сравнивались по 531315 аутосомным снипам. После ряда манипуляций были оставлены 221558 снипов. Были также рассмотрены мтДНК и Y-хромосомные данные из ранее публиковавшихся статей. При анализе центрально-европейские евреи, восточно-европейские евреи и ашкенази принимались эквивалентными. Но поскольку «ашкенази» обычно в литературе принимаются за выходцев из Германии, то авторы, демонстрируя объективность, от термина «ашкенази» отказались. И в самом деле — они настроились на хазарское происхождение европейских евреев, и поэтому термин «ашкенази» им не подходил в принципе. Соответственно, они отказались при рассмотрении и от термина «сефарды».
Поскольку хазарских останков или выявленных потомков у авторов статьи не было, то вместо них использовались биологические материалы европейских и кавказских популяций. Кавказские грузины и армяне были постулированы при исследовании как прото-хазары.
Естественно, как принято в популяционной генетике, измерялись «генетические расстояния между популяциями». В статье, как принято у попгенетиков, вообще не указывалось, у кого брались тесты — у мужчин, у женщин, или у тех и других. Видимо, считается, что это не важно. Но можно полагать, что анализировались геномы тех и других. Правда, мои (А.К.) наблюдения показывают, что в этом случае картину определяют именно мужские снипы, поскольку женские настолько хаотичны и многоплановы (то есть представляют самые широкие вариации), что на суммарную картину влияют совершенно диффузно, то есть не влияют вообще.
Как заглавные в статье рассматривались две гипотезы происхождения европейских евреев — рейнская гипотеза, и хазарская гипотеза. Согласно первой, европейские евреи вышли из малой популяции евреев в Германии, продвинулись на восток и размножились. В этом случае автор ожидает «высокое генетическое сходство между европейскими евреями». Согласно второй, европейские евреи вышли из хазарского каганата (конфедерации славян, скифов, хуннов, булгар, иранцев, аланов и тюрков, обращенных в иудаизм в VIII в.), прошли в Европу и размножились. Из этой хазарской смеси, по мнению автора, вышли хазары, грузины и армяне. Согласно автору, хазарская империя распалась, и после черной чумы в Европе хазары бежали на восток, осели в Польше, Венгрии, и постепенно разошлись по Центральной и Восточной Европе. Поэтому автор ожидает, что в отличие от первой, рейнской гипотезы, хазарская гипотеза должны привести к большему генетическому разнообразию евреев, поскольку они будут происходить от кавказских, европейских, и ближневосточных предков, и разные группы европейских евреев будут отличаться друг от друга по своему происхождению и генетическому разнообразию. То, что разные гаплогруппы евреи могли получить и не от хазар, автору в голову не пришло. Например, гаплогруппа R1a у евреев явно не от хазар, ей примерно 4 тыс. лет, и она, скорее всего, с Ближнего Востока, арийский след.
Правда, для меня как читателя осталось непонятным, почему это немецкие евреи (рейнская гипотеза) оказываются генетически гомогенными (или близкими), если они исходно пришли с Ближнего Востока, будучи рассеянными по Персии, Византии, Ассирии, Средиземноморью, а также пройдя через Европу, чтобы попасть в Германию. Ну да ладно, у автора масса таких условностей, приближений, допущений. Как мы дальше увидим, снявши голову, по волосам не плачут. Иначе говоря, нет смысла к автору придираться по этим мелочам, когда он задачу все равно решил принципиально неправильно. То есть не решил.
Первой задачей исследования автор назначил показать, что рейнская и хазарская гипотезы в самом деле дадут различные предковые картины. И вот здесь начинается акробатика автора. Для начала и для примера он решил выявить генетическую примесь (admixture) между палестинцами и шестью кавказскими и евразийскими популяциями (русские, турки, армяне, грузины, лезгины и адыги), беря как негативный контроль африканцев племени Сан. В общем, начинается уже смешно, поскольку все эти шесть популяций имеют в значительной степени пересекающиеся мужские гаплогруппы. Женщины, как я пояснял выше, здесь не при чем, они дают просто «генетический фон». У всех этих шести популяций есть гаплогруппы R1a, R1b, G2a, Jl, J2 и так далее, в разной, естественно, степени. Но это не важно, в какой степени, все равно все суммируется и усредняется. Популяционная генетика ведь. Так и получилось — то, что назвали f3 статистикой, показало степень «примесности» (admixture) между палестинцами и русскими 0,0011, палестинцами и турками 0,0013, с армянами 0,0019, с грузинами 0,0019, с лезгинами 0,0015, с адыгами 0,0015. Это по автору означает «малую, но значительную примесность». Популяционная генетика. Да непримесности там просто быть не могло, гаплогруппы-то одни. То есть снипы тоже одни, и эти снипы тянут за собой другие, родственные снипы данных популяций.
Но акробатика автора продолжалась. Он показал на армян и грузин, с их примесью с палестинцами 0,0019, и пояснил, что поскольку как армяне, так и грузины разошлись с турками 600 поколений назад (дана ссылка, без какого-либо пояснения), то есть примерно 15—18 тысяч лет назад (???), то в этой примеси доли евреев быть не может, поскольку она была бы недавней. То есть эта примесность «территориальная», а не «семитская». Как написал автор, «сходство между европейскими евреями и кавказцами не вызывается общими семитскими предками».
Кто проникся этими «доводами»? Я — нет. На самом деле доля «примесности» определяется игрой гаплогрупп, среди которых J1 и J2 есть и у евреев, равно как и G2a, и R1a, и R1b, и так далее. У русских J1 и J2 мало, у кавказцев и палестинцев много, вот и выше «примесность». Разделять здесь «территорию» и «семитскость» не имеет большого смысла. Все эти пируэты автора совершенно ничего не дают. Но самое главное, что автор прав — сходство между европейскими евреями и кавказцами не вызывается общими семитскими предками. Но по совершенно другой причине — потому, что их у кавказцев нет (кроме горских евреев), а вот гаплогруппы J1 и J2 есть. И хазары там совершенно ни при чем.
Далее автор перешел к графику «мультикомпонентного анализа», любимому инструменту популяционных генетиков. В нем все популяции перемешиваются, и график показывает, кто к кому льнет. Вот упрощенный (но не очень) характерный пример. Берутся четыре мужских популяции — поляки, русские, казахи и монголы, и строится график — кто к кому ближе. Для этого примера не так важно, что от чего откладывается, но если картина снипов представлена в том или другом виде, то нетрудно догадаться, что поляки и русские, имеющие в основном R1a, окажутся на графике по соседству, а на другом конце графика окажутся казахи и монголы. И попгенетик торжествующе скажет — вот, смотрите, настоящая наука. Кто бы мог подумать, что такой график будет?
Так вот, подобный график построил и автор. На него были положены популяции Ближнего Востока, Кавказа, и европейские евреи.
Теперь мои (А.К.) рассуждения, прежде чем покажем, что получилось у автора. Поскольку гаплогруппы J1 и J2 есть во всех трех популяциях, и много, то окончательный расклад должен определиться балансом гаплогрупп, которых нет или мало в какой-то из трех популяций. Например, у европейских евреев есть много гаплогрупп G2a и R1b, их же много на Кавказе (и к хазарам они никакого отношения не имеют, это древние гаплогруппы), а на Ближнем Востоке их мало. Значит, можно ожидать, что европейские евреи лягут ближе к кавказцам, а не к Ближнему Востоку, хотя финальный ответ даст сложная игра снипов.
Теперь как рассуждал автор: если правильна рейнская гипотеза, то европейские евреи лягут ближе к Ближнему Востоку, а если правильна хазарская — то ближе к Кавказу.
Мы уже, впрочем, видим, что хазары здесь опять ни при чем. Потому что на Кавказе есть много Jl, J2, G2a, R1b. И R1a тоже может какую-то роль сыграть, этой гаплогруппы на Кавказе немного, но она есть и у европейских евреев.
Так и получилось. Более 70% европейских евреев и почти все восточно-европейские евреи легли на графике ближе к кавказцам, и именно к грузинам, армянам, и азербайджанским евреям. Вот, восклицает автор, это — сильное доказательство хазарской гипотезы. Более того, восклицает автор, миграции кавказцев в Европу до времен хазарской империи неизвестны, значит, это хазары мигрировали, и принесли кавказские снипы вместе с евреями в Европу.
Это, конечно, смеху подобно. Евреи сами по себе принесли свои гаплогруппы Jl, J2, R1a, G2a, R1b в Европу (и получили от других европейцев, как R1a они получили от древних ариев на Ближнем Востоке примерно 4000 лет назад), и эти же гаплогруппы испокон веков, а именно не менее 6 тыс. лет назад, были на Кавказе. Поэтому кавказцам никуда и ходить не надо, у них все это было с древнейших времен. И без хазар. Параллельно с евреями.
Технически это не исключает наличия евреев на Кавказе, в том числе и как хазар. Но это ничего не доказывает, как наивно считает автор. Методология его в принципе неверна.
Опять и опять автор повторяет, что рейнская гипотеза не может объяснить геномное сходство евреев с кавказцами, а хазарская — может. Опять нам здесь приходится говорить, что к хазарам это не имеет никакого отношения. Эти гаплогруппы попали на Кавказ с Ближнего Востока задолго до евреев, как попали они к евреям тоже оттуда, потому и глубинное сходство. Но популяционные генетики в исторических терминах не мыслят. Они смотрят на то, что ЕСТЬ СЕЙЧАС. И отсюда уже фантазируют на предмет истории.
Впрочем — цитирую автора: «Хотя европейские евреи образуют кластер с кавказскими популяциями, восточно- и центрально-европейские евреи делят большой кластер с западноевропейскими и ближневосточными популяциями». Ну естественно, они и должны делить «большой кластер», гаплогруппы опять те же самые. Нет там «или — или», все определяется, повторяю, игрой долей гаплотипов и снипов в популяциях, и мозаика этого может быть самой разной. Но поскольку автор вводит много степеней свободы в интерпретации, ими можно объяснить что угодно. Сходство снипов евреев с Западной Европой объясняется уже вводом в игру «греко-романских евреев», там же «израильские прото-иудеи», и «месопотамские евреи».
Все остальные акробатические этюды в статьи того же свойства — в основе их сходство гаплогрупп кавказцев и европейских евреев, называть их ашкенази или не называть. Автор применяет как меру сходства так называемый IBD (identity by descent), при котором сопоставляют сходство средних геномных участков по их «длине». Оказалось, что и в этом случае длина этого усредненного сходного участка между европейскими евреями и кавказцами 9,5 сМ, а между теми же евреями и палестинцами только 5,5 сМ. Опять, автор считает, что тому причиной хазары, в то время как это — сходство гаплогрупп между ашкенази и кавказцами. Опять хазары здесь ни при чем.
И после этого — длинное обсуждение «результатов» с предполагаемыми маршрутами и направлениями передвижения хазар, что якобы вытекает из исследования.
В заключение я хотел бы подчеркнуть два «момента». Во-первых, этот разбор статьи вовсе не означает, что хазар как таковых не было. Но так, как искал автор статьи, их искать нельзя. Во-вторых, я вовсе не утверждаю, что игрой гаплогрупп можно полностью заменить игру геномов. Но это то, что надо специально проверять и изучать, в какой степени заменить можно, и в каких случаях. Дело в том, что я уже не раз сталкиваюсь с тем, что гаплогруппы Y-хромосомы играют определенную и значительную роль в геномном анализе. Там есть какая-то тайна, почему столь маленькая хромосома со своей системой снипов как паровозик тащит за собой сотни тысяч снипов со всего генома. Ясно, что эта корреляция не случайна, за ней что-то кроется. Я опять напомню, как снипы гаплогруппы Р и ее потомков, гаплогруппы Q, R1a, R1b, потащили за собой несколько процентов якобы «америндского компонента» (гаплогруппа P→Q), обнаруживаемого в Западной (гаплогруппа P→R→R1→R1b) и в Восточной Европе (P→R→Rl→R1a). И это те же генетики в своей бесконечной мудрости назвали «долей генома американских индейцев в ирландцах и русских». А это, напротив, европеоидные снипы в американских индейцах.
Вот такая популяционная генетика.
Для начала — несколько вводных положений.
Первое — изучение генома человека чрезвычайно важно как для развития биомедицины, в первую очередь — «персонализованной медицины», так и для изучения происхождения человека, его эволюции, для ДНК-генеалогических исследований как популяций, так и конкретных семей, их генеалогических линий.
Второе — изучение генома в значительной степени базируется на мутациях в хромосомах ДНК (а также в митохондриальных ДНК, которые тоже часть общего генома).
Третье — изучение генома по мутациям в ДНК может проводиться на разных уровнях приближений, а именно на разных фрагментов ДНК — от малых (например, фрагментах Y-хромосомы или любых других хромосом), так и по всему полному геному человека как индивидуально, так и популяций человека, вплоть до всего (в далекой перспективе) населения Земли.
Это все важные положения, и не о них мое ироническое, если не сказать саркастическое название этого постановочного полемического рассказа. Любое грандиозное по важности исследование, любой передовой подход можно свести на уровень профанации, если не следовать вполне общепринятым научным принципам. К сожалению, эта профанация наблюдается, причем широко, в «популяционной генетике» человека. Почему именно в этом направлении, вполне, казалось бы, безобидном и ориентированном на конкретные исследования современных популяций? Что плохого в том, что популяционные генетики изучают определенные популяции, этносы, наследственные линии, и выявляют в них генетические аберрации, свойственные одним популяциям и совсем несвойственные другим?
Ничего плохого в этом нет, это, напротив, замечательно. Правда, плохое там может быть, когда имеет место утечка информации персонального характера, и страховые компании отказывают в страховке, или поднимают цену для конкретных людей, потому что они находятся в группе «повышенного риска». Хотя и в этом некоторые находят правильную позицию страховых компаний, что повышенный риск должен повышенно оплачиваться, тем более, когда рискующие знали, на что идут, выходя замуж или женясь на человеке из той же группы повышенного риска, возводя риск в квадрат, а оплачивать это в случае патологии должны другие люди. Ведь страховка профессиональных спортсменов намного выше, чем «обычных» людей, и страховка гоночных автомобилей намного выше, чем обычных. Как бы там ни было, а популяционные генетики делают хорошее и важное дело, выявляя связь генотипа и фенотипа, в общей формулировке. Собственно, это и есть главная задача популяционной генетики.
Проблема начинается тогда, когда популяционные генетики берутся не за свое дело, и выходят за пределы своей профессиональной компетентности. Например, когда берутся за ДНК-генеалогию с ее количественным математическим аппаратом, по сложности превышающем обычный уровень популяционных генетиков. Они, конечно, могут попытаться научиться, но они не хотят. Не хотят принципиально. В итоге получаются не только сплошные конфузы, но и полная дискредитация своей науки. Критический уровень этой дискредитации уже давно зашкаливает.
Сначала, в конце 1990-х — начале 2000-х, это были «датировки», которые брались «по понятиям», а на самом деле совершенно «с потолка», без всяких обоснований. Соответствующие цитаты я уже давал предостаточно в статьях, где это обсуждал. Могу только напомнить про «R1b в Европе 30 тыс. лет назад», про «украинскую гаплогруппу R1a, которая спасалась в украинском же убежище в ходе ледникового периода», про «выход современного человека из Африки 70 тыс. лет назад», и так далее. Никаких расчетов на самом деле не проводилось, да их и быть не могло. Это — стиль популяционной генетики. Потом, в середине 2000-х, взял свое начало «метод Животовского» с его «популяционными скоростями», которые совершенно некритично принимались в любом случае, для любых гаплотипов, для любых маркеров, в любом временном интервале. Самое прискорбное — что никто из «популяционных генетиков» и вопросы не задавал, применяли совершенно не думая и не осознавая. В итоге — обычное завышение результатов расчетов на 200—300% (!). Откуда это, в чем причины? Причины — в полном отсутствии нормальной научной школы, в которой эти несуразицы вскрываются на первом же научном семинаре. Причины — в полном контроле «популяционными генетиками» профильных научных журналов. В подборе рецензентов, которые тоже полностью контролируются, а потом привыкают. Или привыкли с самого начала.
Шокирующих примеров слишком много, чтобы все публиковать. Один из краеугольных камней «попгенетиков», которые устремляются в «исторические науки» — это исходить из статической картины гаплогрупп: то, что наблюдается в настоящее время, то же якобы было всегда. Оттуда и возникли те самые R1b давностью в Европе 30 тыс. лет — они же есть в Европе сейчас, значит, были всегда. Ну а 30 тыс. лет — из вежливости, сверху уже неандертальцы подпирают. Так и сейчас, уже в 2013 году, нашли Rlb-L51 в центре Европы — значит, там и образовались. Нашли R1b в Анатолии — значит, там и образовались, оттуда и пришли. О том, что они и в Сибири есть — авторы и не знают, их карта в статье на Турции заканчивается, восточнее и не смотрели. Популяционная генетика. Нашли стоянки в Турции давностью 10 тыс. лет назад — значит R1b, как будто других гаплогрупп нет, например, G, Е, J, R1a, F, К и так далее.
Вот такая популяционная генетика.
Еще один показательный пример — некто Диенекес Понтикос, ведущий свой собственный блог «по антропологии», решил, что констант скоростей мутаций ДНК нет, точнее, что они невоспроизводимы и меняются от системы к системе. Почем он так решил? Да потому что в них не разбирается. И объявил о запрете рассмотрения их на своем блоге. Так и объявил — ввожу «бан» на скорости мутаций. История забавная.
И вот тот же Д. Понтикос сейчас занялся геномом, точнее, сериями мутаций в нем. И в соответствии с ментальностью многих попгенетиков, стал применять принцип — «что вижу, о том пою». То есть беру картину мутаций в Польше — называю ее «польской картиной». Неважно, что она такая же, как в Беларуси или в Литве — пусть будет «польской». Вижу картину у американских индейцев, неважно, что они пришли из Сибири со своей сибирской гаплогруппой Q, пусть будет каноническая картина «американских индейцев». Причем американских индейцев в Сибири. Или американских индейцев во Франции. Или в Ирландии. Или в России. Отныне они все будут в своей части «американские индейцы». Узнаете «популяционную генетику»?
И вот такие люди закладывают основы новой дисциплины, при полном попустительстве остальных. И эти остальные чешут голову и гадают — откуда в Ирландии американские индейцы? Но раз Понтикос сказал, значит, так оно и есть. «Доктор сказал в морг, значит в морг».
Возвращаемся к началу нашего повествования.
Уже довольно давно я с определенным подозрением слежу за упражнениями Д. Понтикоса со снипами по всему геному. Понтикос, по своему незнанию и непониманию, сначала объявлял, что огромные погрешности в расчетах времен до общих предков делают эти расчеты бессмысленными. Но оказалось, что он в этих расчетах ничего не понимает. Мне препираться с ним надоело, и я задал ему конкретный вопрос — есть сто 67-маркерных гаплотипов, в которых суммарно 2000 мутаций. Когда жил общий предок этих ста гаплотипов, и с какой погрешностью определяется эта величина? Понтикос не ответил и предпочел ретироваться. Больше со мной он этот вопрос не поднимал.
Ответ на самом деле очень прост — 5000 лет назад с погрешностью плюс-минус 510 лет. При этом нужно оговорить два условия — что все эти гаплотипы происходят от одного общего предка, и константа скорости мутации определена с точностью плюс-минус 10%. Если первое условие не выполняется, гаплотипы делятся на ветви, нет проблем. Второе условие обычно завышено, и погрешность на самом деле часто оказывается меньшей, ну да ладно, пусть будет для страховки. Лучше погрешность завышать, чем уменьшать.
Но Понтикос не успокоился, и вскоре объявил о том, что расчеты времен до общего предка не имеют смысла, потому что зависят от того, какие маркеры используют для расчетов. Его в этом отношении окрылила статья Busby et al (2011), которая оказалась предельно безграмотной (или запредельно безграмотной), хотя и опубликована в журнале Английского Королевского общества. Busby и соавторы взяли несколько «скоростей мутации» для отдельных маркеров, и нашли, что для разных маркеров получаются разные времена для общих предков. Эти «скорости мутаций» они взяли из сопоставления величин аллелей в примерно 1750 парах отец-сын. При этом английские авторы оказались настолько далекими от понимания простых вещей, что не поняли, что эти «скорости мутации» просто нельзя рассматривать для подобных расчетов. Например, в 1750 парах DYS393 мутировал три раза, а DYS390 — два раза. То есть DYS393 оказался более «быстрым», чем DYS390 (!). И авторы английской статьи, а вслед за ними и Понтикос, так и посчитали, что скорость 393-го более высокая, чем 390-го. И это и использовали в расчетах! Между тем, любой, кто хоть однажды видел серии гаплотипов, видел, что 393-й мутирует крайне редко, а 390-й строчит как из пулемета. То есть и английские авторы, и Понтикос не имеют о таких вещах ни малейшего понятия. Они не имеют понятия и о том, что 2 и 3 мутации в 1750 парах отец-сын — это никакая не статистика, там могла легко быть и 1 мутация, и 4—5 мутаций. Нельзя на таком уровне частоты событий вообще что-то рассчитывать. Это все равно, что бросить монету три раза, и на этом основании рассчитывать вероятность выпадения орла или решки. И на этом основании и английские авторы, и Понтикос делают выводы, что по мутациям ничего считать нельзя! Более того, Понтикос объявил на своем сайте, что я уже упомянул выше, что отныне он делает «бан» на расчеты ДНК-генеалогии и больше ничего на эту тему не помещает.
Следующим действием было его увлечение расчетами возраста гаплогрупп на основании числа SNP в гаплогруппах и субкладах по данным проекта «1000 геномов». Как он считал — он поначалу не показал, и просто опубликовал список «возрастов» по гаплогруппам. Три четверти из них совпадали с нашими датировками, опубликованными совместно с И.Л. Рожанским в нашей статье про Африку в Advances in Anthropology за полгода до того. Ссылки на нас он, понятное дело, не дал, но отметил, что считать по гаплотипам — это неправильно, надо считать по снипам. Я выступил на форуме RootsWeb, и задал ему вопрос — если он получает такие же цифры, как у нас, более того, подозрительно такие же, как он может говорить, что по снипам считать — правильно, а по гаплотипам — неправильно? Он стал скандалить, что это у нас неправильно, и стал приводить цифры по той четверти случаев, в чем у нас разница. Там, где совпадало, он умолчал. Но это было неудивительно, его натуру я уже знал.
Но произошла неожиданность — выступил другой человек, и сказал, что он использовал те же снипы из проекта «1000 геномов», и тот же в принципе метод расчета, и получил совершенно другие данные, чем у Понтикоса. В частности, как видно, Понтикос не делил полученные датировки на 2, хотя он получал не возрасты гаплогрупп, а РАССТОЯНИЯ между ними. Понтикос опять ретировался и замолчал. Тут уже вышел я и потребовал объяснений. Он же до того объявил, что мы считаем неправильно, а он — правильно. Мое требование пришлось повторять трижды, Понтикос молчал. Потом он вышел и повинился, что на 2 он действительно не делил, но во всем виноват проект «1000 геномов», потому что они выдают неправильные данные, и вообще данных недостаточно для подобных замечательных расчетов, какие делает Понтикос. Короче, он дезавуирует, то есть снимает свои данные как действительно неправильные, потому что по снипам получается что угодно. Извинения мне он, конечно, не принес.
К чему я это все описываю? А для того, чтобы показать, что объявления и расчеты Понтикоса гроша ломаного не стоят. И это уже переходит в новое увлечение Понтикоса, под названием ADMIXTURE.
И вот началась эта следующая эпопея — популяционные генетики добрались до данных генома, и стали сравнивать сотни тысяч снипов по популяциям. И Понтикос там занял одну из ведущих ролей. Но как истый популяционный генетик по духу, он не понял всей сложности картины. Дело в том, что считают по геномам как мужчин, так и женщин, совместно, не разделяя. Все это образует, естественно, облака снипов, которые только условно могут быть приписаны конкретным регионам, и там они пересекаются, накладываются поперек регионов. Но Понтикос сразу же стал приписывать этим облакам этнические названия, чисто в попгенетическом духе. Более того, даже там, где он приписывал этим облакам (которые он усредняет до якобы точек) названия континентов, или популяциям континентов, эти названия настолько упрощены, что стали ошибочными. На форуме «Родство» польский визитер уже сообщал, что у него 95,6% «польских снипов». То, что польские, литовские, белорусские, да и русские дают примерно такую же картину, он не знал. Иначе говоря, это почти все равно, что назвать «польской» гаплогруппу R1a, и продолжать писать, что у уйгуров, например, 30% польского генома. Что из этого получается — можно сказать коротко. Профанация.
Вот — конкретный пример, который имел место совсем недавно. Мой знакомый ирландец, с которым у меня в печати находится статья про историю субклада Rlb-M222 в Европе, выставил сообщение на RootsWeb с недоуменным вопросом. Он скачал программу, которую составил Понтикос на своем сайте, вставил свои (и родителей) данные по геному, и с удивлением обнаружил, что он и его родители почти на 10% — американские индейцы. Он выставил свои данные и с удивлением заметил, что его родители и он сам — «коренные ирландцы» из Ирландии, и никаких индейцев у них в роду отродясь не было. Вот что он выставил:
«Здесь мои родители и я — все ирландцы, никаких предков среди американских индейцев.
Есть здесь еще с предками от американских индейцев?
Paul».
Поднялась дискуссия, в которой обнаружилось, что у всех заметное присутствие доли американских индейцев.
Кстати, никто не обратил внимания, что в геноме матери африканцев вообще нет, а у самого Поля почему-то их больше, чем у отца. На самом деле это все на уровне «шума». Нет в нас мутаций африканцев, не выходили наши предки из Африки. Из общего предка с шимпанзе — масса мутаций, а от африканцев — нет.
Некоторые подняли вопрос, что в программе с американскими индейцами что-то не так. Другие защищали, что раз Понтикос сказал, то так тому и быть (напоминаю: «доктор сказал в морг, значит в морг»), и стали придумывать гипотезы одни фантастичнее других, и абсурднее третьих.
А ларчик на самом деле просто открывался. Понтикос в своем попгенетическом рвении, вместо того, чтобы обозначить определенные закономерности цифрами или индексами, назвал их просто с потолка. Перевод: четыре предковых популяции появились на этом уровне разрешения, которые я назвал: европейская, азиатская, африканская, америндская. Имена не важны, и вы можете заменить их какими хотите.
Мой комментарий: если имена не важны, то какого дьявола их вообще называть так? Три дня эти чудаки на RootsWeb спорили о том, откуда у них в предках американские индейцы, а это вообще, судя по всему, сводная гаплогруппа P. Она дала гаплогруппы Q, R→R1→R1a+Rlb, а ирландцы — это и есть гаплогруппа R1b на 90+%, и наш Paul — Rlb-M222. Q ушла в Америку, и ее имеют многие американские индейцы. Вот и вся разгадка. Если геном имеет массу снипов от общего предка с шимпанзе 6 миллионов лет назад, то как же ему не иметь снипов от гаплогруппы P, всего 40 тыс. лет назад? Они, эти снипы, и ушли как в Америку, к индейцам, так и в Ирландию, так и ко всем R1b, так и в Восточную Европу (и не только), ко всем R1a.
И действительно, у русских примерно столько же вклада «американских индейцев», о чем Понтикос старательно (и тупо) доложил:
Украинцы — 8,5% америндов.
Поляки — 8,2% америндов.
Литовцы — 9,1% америндов.
Русские — 11,7% америндов.
Славяне в целом — 9,1% америндов.
Понтикос так и пишет: «For example, HGDP Russians have 11.7% of Amerindian component». To есть то, что это понятие «америнды» — он ввел чисто условно, он уже не вспоминает. Раз подхватили, зачем менять, не так ли?
Надеюсь, понятнее теперь, что эти цифры и этносы у Понтикоса на самом деле никакой смысловой нагрузки не несут?
Ущербность этого подхода, повторяю, и в том, что все эти облака усредняются до точки в каждом случае, и эти точки и приводятся с точностью до долей процента.
На самом деле эти данные, если их обрабатывать правильно, несут вполне ценную информацию. Обычно рассматривают сотни тысяч снипов у каждого человека, и они, ясное дело, есть объективная реальность. В эти снипы входят снипы и шимпанзе, точнее, общего предка шимпанзе и человека (но их осталось и в шимпанзе и в нас более 90%), и все снипы наших гаплогрупп и субкладов Y-хромосомы (которых у женщин нет, но все равно усредняется между полами), и снипы наших отцов и матерей, что отражает причудливую картину их взаимного выбора, а поскольку часто мужчины в этносах выбирают женщин из того же этноса, и наоборот, то картины в целом по этносам расходятся. Примерно так: если взять группу русских (мужчин и женщин) и группу монголов-монголок, то у них будут — у тех и у других — масса снипов шимпанзе, снипы альфа-гаплогруппы, снипы бета-гаплогруппы (пока все примерно одно и то же), но потом пошла разница — у монголов в немалой степени гаплогруппы С, Q, R1a у мужчин (R1a как древние, так и недавние вливания от России и СССР), и соответствующие мтДНК от женщин, которых у монголок весь иконостас, и у русских R1a, I1, I2, N1c1, и еще куча более минорных снипов, плюс гаплогруппа H (преимущественно) от женщин, плюс вся палитра других женских гаплогрупп, причем у русских это варьируется по всему горизонту. И вот эта чудовищная каша усредняется «попгенетиками» и «геногеографами» в одну точку! И это называется «русский геном» и «монгольский геном», и вот они-то сравниваются с точностью до долей процента.
Все это, конечно, полная чушь. Это я — о точке и о долях процента. На самом деле этот «русский геном» пересекается со всей Восточной Европой, и его не в точку нужно стягивать, а найти другой характер представления. А именно, в виде соответствующих облаков, растянутых неравномерно в разные стороны.
Но и в этом случае различия все равно будут между русскими и монголами. Качественно и как-то полуколичественно его можно рассматривать, но не в виде профанации, как это делает Понтикос. Более того, это рассмотрение — если правильно — надо проводить не на выбранных маленьких фрагментах, а действительно по всему геному. На маленьких фрагментах будут вылезать отдельные особенности — то присущие в основном, например, гаплогруппам Y-I2 и мтДНК-Н, то кому-то еще. И это еще будет зависеть от разрешения, которые и обозначают индексами К=4, К=8 и другими. То есть берут маленький фрагмент генома, да еще с малым (или бо́льшим) разрешением, стягивают в точку, и все равно получают в целом ерунду. Но для коммерции годится. Годятся для коммерции и вот такие, в частности, «открытия» того же Понтикоса.
Перевод: «Интересно то, что европейская популяция показывает присутствие американских индейцев, что показывает f-статистика, и она же показывает присутствие компонента с Сардинии».
Как видим, Понтикос уже забыл, что названия им придуманы как попало, и уже придает им абсолютные значения.
Про Сардинию Понтикос уже вошел в состояние экзальтации. Он придает Сардинии некую пра-европейскую значимость, на основании, конечно, этой ерунды с «геномом», который анализирует как хочет. Пример — он трубил по всему свету, что Отци, «ледовый человек», имел геном «Сардинии». Однако, только что опубликована статья, что Отци — никакая не Сардиния, а типичная Центральная Европа. Ну, и что с «трубил по всему свету» делать будем?
Понтикос, с его страстным желанием сенсаций, каждый раз наступает на одни и те же грабли. Впрочем, фарс продолжается. Теперь тем же занялся некий российский Веренич, а именно — тоже насчитывает «польскую компоненту», пользуясь подходом своего гуру Понтикоса.
Я не проводил детальный разбор все этих admixture подходов, но беглое их рассмотрение показывает, что — как ни странно — картина в первую очередь диктуется мужскими гаплогруппами (Y-хромосомы), а женские гаплогруппы (мтДНК) практически не имеют значения. На первый взгляд это абсурд, так как примерно половина участников в расчете admixtures — это женщины. У них мужской хромосомы нет. Но если подумать, то описанный феномен не исключен. Я уже об этом писал в свое время. Женские гаплогруппы хаотичны, они не образуют четкой картины, и они накладываются на значительно более упорядоченные мужские гаплогруппы. Более того, они часто образуют пары с мужскими гаплогруппами. В итоге имеем корреляции между частью мужских и женских гаплогрупп, и хаотичность женских у остальных. Женских гаплогрупп зачастую не видно, они прозрачны в своей размазанности, как прозрачен винт самолета при его вращении.
На эту мысль меня навело беглое рассмотрение картин мутаций в геноме при 50:50 наличии мужчин и женщин. Оказалось, что на вид эти раскрашенные картинки геномов практически совпадают с картиной мужских гаплогрупп. У русских — половина одного цвета (это R), который одинаков с поляками, как и должно быть, и он совпадает с тремя четвертями у французов и 90% англичан и ирландцев, у которых столько и есть R. При том разрешении R1a и R1b не разделяются. Далее, у русских примерно 1/6 другого цвета, которого три четверти у финнов. Это, ясно, гаплогруппа N. Очень мало желтого цвета, который захлестывает монголов. Это — С или Q, надо разбираться. Совсем нет черного цвета, которого полно в Африке. Сейчас мы увидели (см. выше), что у ирландцев, русских и прочих европейцев около 10% чего-то, что доминирует у америндов. Это явно связано с гаплогруппой Р или Q.
В общем, в этом нужно разбираться, и странно, что никто из тех, кто упражняется в admixture, это не заметил. Я призываю тех, кому интересно, заняться и посмотреть, насколько это действительно контролируется мужскими гаплогруппами. В принципе, данные по геному — объективные данные, дело в интерпретации.
Ниже — мои слова и призывы к молодым специалистам и любителям на сайте «Родство» при обсуждение данных по геному:
— Там много в чем надо разбираться, но от этого никуда не деться. Формируется новый и мощный подход в генетике, основанный на объективных данных по набору мутаций в хромосомах и во всем геноме. Вопрос — как эти данные трансформировать в хронологические показатели, причем выявить их отношение к регионам, древним миграциям, и провести параллели-корреляции с гаплогруппами-гаплотипами и их хронологией.
При этом либо наши данные нужно будет подправлять на основании полного (или частичного) генома, но в этом надо будет надежно убедиться (наука есть наука, и такая вероятность всегда есть), либо, напротив, геном полностью подтвердит наши подходы и данные. Тогда это будет сильным козырем, против которого не возразить. Если мы этого не сделаем, мы отстанем, чего допустить никак нельзя.
Проблема в том, что в это уже полезли попгенетики, и как им генетически присуще, начинают это дело портить, превращать в обычную для них свалку и помойку. Так что опять, к сожалению, прогрессивно формируется новое поле для схватки, для битвы, для борьбы науки с нахрапистым гаданием и жонглированием. Это у попгенетиков системное.
Поэтому принцип все тот же — если не мы, то кто же? Мы это видим в ДНК-генеалогии гаплотипов, придется увидеть и в ДНК-генеалогии генома. Это же та же ДНК-генеалогия.
Так что у меня призыв к коллегам, особенно к коллегам молодым (но не только) — засучить рукава и разобраться в новом направлении. Базы данных по геному прогрессивно растут, они доступны, программы по анализу этих данных уже есть и тоже их число нарастает. Надо понять суть всех этих К=4 и прочих, убрать эту этничность, с которой носятся попгенетики, забыть про все эти «польские компоненты», которые здесь ни к селу, ни к городу и имеют плохой коммерческий привкус (или откровенное шарлатанство, чем сейчас и занимается Веренич, судя по описаниям канадского гостя), и посмотреть, насколько это привязывается к гаплогруппам и субкладам Y-хромосомы.
Дело не в том, входят ли в сотни тысяч снипов в данном подходе конкретные снипы гаплогрупп. Эти конкретные несколько снипов все равно растворятся в сотнях тысяч других. Дело в том, что эти несколько снипов гаплогруппы формируют те самые сотни тысяч анализируемых снипов, или десятки тысяч в них. Все это в целом образует общую картину, в которой корни популяции давностью десятки тысяч лет тащат за собой всю картину, точнее, ее значительную часть, включая и женскую половину.
А женскую — потому, что мы женимся в основном не на негритянках и не на австралийских аборигенках, и не на монголках, поэтому их мутаций в нашем геноме мало. Иначе говоря, это не статистическая каша, а весьма структурированный набор снипов в геноме. Фон, естественно, есть, и сильный, от тех же предков с шимпанзе, но его либо отфильтровывают при анализе, либо учитывают другим образом.
Именно потому должна быть корреляция с гаплогруппами Y-хромосомы, или, если ее нет, то это надо четко показать. Думаю, что она будет. Вопрос, в какой степени.
Классическая задача популяционной генетики — это нахождение связи (корреляции) между генотипом и фенотипом. Это опять не имеет отношение к ДНК-генеалогии. Поэтому я все время и повторяю, что попгенетики лезут не в свое дело, причем при полном отсутствии знаний и квалификации в этом не своем деле.
Опять же классический вопрос попгенетики — это выявление набора генов, являющихся причиной наследственных заболеваний. Опять к ДНК-генеалогии это не имеет отношения. Вопрос важный, и любимым объектом у попгенетиков являются, например, евреи с их букетом характерных наследственных болезней. А у киргизов, например, таких нет. Вот описание этого и есть поле попгенетики. Оттого и их любимые графики F, в которых все гаплогруппы смешиваются в кучу, и вообще не рассматриваются. Потому что они в генетике (попгенетике) не нужны.
Похоже, этот же подход сейчас используют при анализе генома. И слава Богу, пусть используют, в медицине это важно. Только это опять же не ДНК-генеалогия.
Но я о другом. Эти данные и коллективный анализ мутаций можно явно рассматривать и применять в ДНК-генеалогии. И это — очень интересное поле науки. Просто нужно по-другому смотреть на те же вещи.
Вот еще пример, в котором ясно проглядываются гаплогруппы. Это — из дискуссии на англоязычном форуме, в которой опять рассматривают мутации в геноме, но гаплогруппы не замечают, и описывают некий «атлантически-средиземноморский типаж» в геноме.
На самом деле этот «типаж», или «компонента» — в значительной степени гаплогруппа R1b. Этот геномный типаж доминирует у басков, сардинцев, французов и англичан, и довольно высок у немцев. Затем значительно падает у поляков, и становится совсем малым у литовцев и финнов. Именно так проявляется гаплогруппа R1b. Ну, и где здесь независимый вклад мтДНК, и в чем это выражается?
Далее, то, что называют «северно-европейский» геномный типаж, это явно в основном гаплогруппа N (N1c1), возможно, в комбинации с H. Этот типаж отсутствует в Сардинии, его совсем немного у французских басков, увеличивается при движении от Франции в Британию и Германию, достигает значительных величин у поляков, и доминирует в Литве и Финляндии.
Ясно, что мужские гаплогруппы в целом могут определять картину снип-мутаций в геноме, вопрос только, в какой степени и в каких ситуациях. Непредвзятые исследования это покажут.
А пока — вот что только что появилось в связи с новой системой тестирования Geno 2.0, Система исключительно маркертирована, и, хочется надеяться, что-то хорошее принесет для идентификации снипов. И вот пошли первые результаты. Оказывается, в проект уже влезли «геногеографы» и началось то, о чем я писал выше. Они подразделили ожидаемые данные по регионам, и стали приписывать, кто к какому региону принадлежит, в процентах.
Вот, например, «портрет русского» по их данным:
На 51% «северные европейцы».
На 18% юго-западные азиаты.
На 25% средиземноморцы.
На 4% северо-восточные азиаты.
Кто-либо что понял?
Ну так вот, «портрет финна»:
На 57% «северные европейцы».
На 17% юго-западные азиаты.
На 17% средиземноморцы.
На 7% северо-восточные азиаты.
Если не все еще поняли, то мы — это финны.
Откуда финны на 17% средиземноморцы — это тоже на трезвую голову не понять.
Немного проясняет вопрос то, что это все вычислялось по «референсной выборке» геномов из русских и финнов. Откуда эти «референсные выборки» геномов брались, например, у русских — тоже надо разбираться, как и с тем, сколько в этой «референсной выборке» было геномов. Не исключено, что было совсем немного, причем с финно-угорской территории где-нибудь севернее Пскова, где N1c1 более 30%.
Вспоминается, что на Форуме еще давно негодовали, что попгенетики представили в международный консорциум в качестве «русских» представителей угро-финнов из какого-то карельского региона, и теперь это идет как «стандартная русская выборка». Если это действительно так, то пиши пропало. Никакие Geno 2.0 не помогут.
Попгенетика продолжает маршировать.
До поры, до времени.